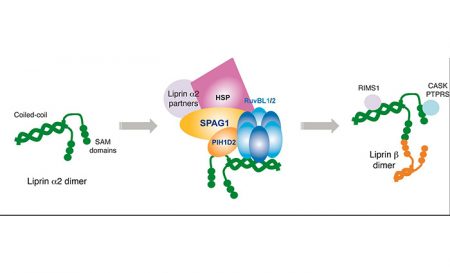
Dans leur étude publiée dans Nature Communications, Chloé Maurizy et ses collègues (équipe d’E. Bertrand et trois autres équipes dans le cadre du consortium ANR snoRNPASSEMBLY) ont découvert une nouvelle co-chaperonne, appelée R2SP. L’assemblage fonctionnel des complexes macromoléculaires essentiels à la viabilité cellulaire nécessite des machineries dédiées telles que le R2TP déjà connu, fonctionnant avec Hsp90. R2TP est composé d’un hétérodimère RPAP3-PIH1D1, associé à un hétéro-hexamère d’ATPase RUVBL1-RUVBL2, qui porte vraisemblablement l’activité chaperonne. Les chercheurs viennent de résoudre la structure tridimensionnelle par RMN du domaine C-terminal de RPAP3 et de montrer que ce domaine s’associe in vivo et in vitro avec le complexe RUVBL1/2. Des analyses systématiques d’interaction montrent qu’une protéine paralogue à RPAP3, nommée SPAG1, lie directement PIH1D2 (protéine paralogue à PIH1D1) et les RUVBL1/2 pour former un nouveau complexe de type R2TP, appelé R2SP. L’équipe a notamment montré qu’elle est requise pour la stabilisation d’une protéine signal, la liprine, et pour son assemblage avec ses partenaires, ces complexes agissant dans le cerveau et les testicules.
Ces résultats permettent de mieux comprendre les mécanismes moléculaires d’action du système chaperon-cochaperon HSP90/70-R2TP et -R2SP, dans le repliement et l’assemblage quaternaire de protéines et complexes macromoléculaires clients. Un dysfonctionnement dans ces étapes est à l’origine de plusieurs pathologies humaines, neurodégénératives comme Alzheimer, Parkinson et l’atrophie spinale musculaire, ou rares comme la dyskératose congénitale et la rétinite pigmentaire, et enfin les ribosomopathies.