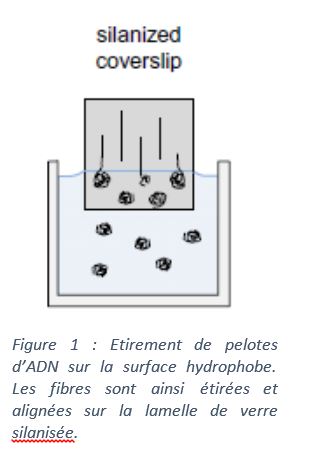
Le contexte
Le peignage moléculaire de l’ADN est une méthode biophysique développée à l’Ecole Normale Supérieure de Paris puis à l’Institut Pasteur (Bensimon et al., 1994; Michalet et al., 1997) permettant d’aligner avec un facteur d’étirement constant (~2 kb/µm) de longues molécules d’ADN (jusqu’à 5 Mb) sur des lamelles de verre silanisées. Cette technique fournit une méthode quantitative pour suivre l’activation et la progression des fourches de réplication à l’échelle de la molécule individuelle, après incorporation in vivo d’analogues de la thymidine dans l’ADN nouvellement synthétisé (Lengronne et al., 2001). Contrairement aux approches basées sur les populations (Gels 2D, ChIP/ChIP, ChIP/seq…), cette technique prend en compte la nature stochastique de l’activation des origines et révèle la dynamique de réplication au sein de cellules individuelles. Les valeurs obtenues ne sont pas moyennées sur la population et permettent donc de détecter des altérations subtiles de la réplication qui sont invisibles par les autres techniques d’analyse de la réplication.
Comment ?
Le peignage moléculaire a deux applications principales : i) l’analyse de microremaniements chromosomiques (délétions, inversions, amplifications de 50 à 500 kb) ; ii) l’étude fine de la dynamique de réplication des chromosomes eucaryotes. Pour cette dernière application, les cellules en cours de réplication sont pulsées avec des analogues de la thymidine (IdU, CldU), l’ADN génomique est préparé puis déposé (peigné) sur les lamelles silanisées. L’incorporation des analogues est détectée à l’aide d’anticorps spécifiques couplés à des fluorophores distincts, le long des molécules individuelles d’ADN pouvant atteindre 5 Mb de long (Figure 3).
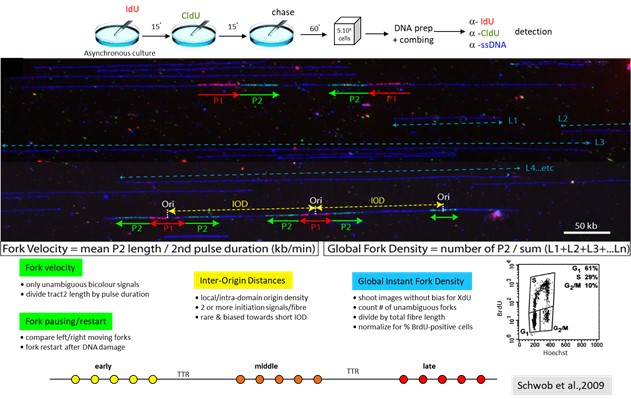
Plusieurs paramètres importants pour comprendre les anomalies de la réplication peuvent être obtenus à partir de ces images :
– la vitesse individuelle des fourches de réplication (FV)
– l’asymétrie de progression des fourches à droite et gauche des origines (FA)
– les distances inter-origines (IOD)
– la densité globale instantanée en fourches de réplication (GIFD)
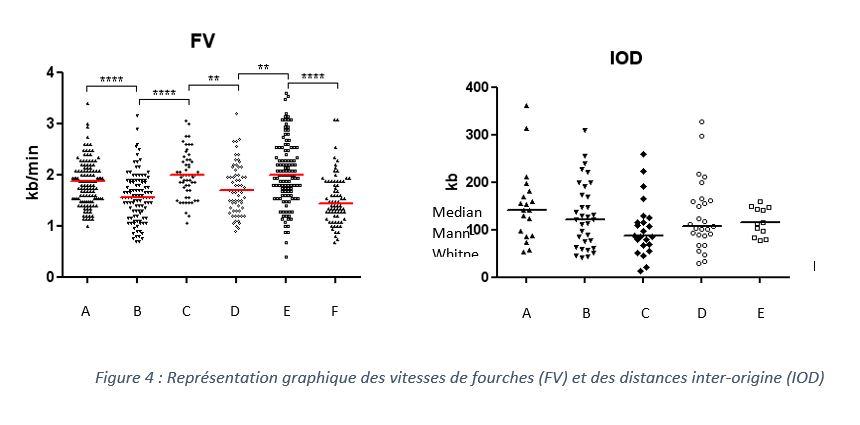 Ces mesures permettent de détecter l’existence d’un stress réplicatif lié à une diminution des pools de nucléotides ou une inhibition des DNA polymérases (FV réduite), à des dommages à l’ADN (FA élevée), une diminution locale ou générale du licensing ou de l’activation des origines (IOD élevée, GIFD réduite)(Bialic et al., 2015).
Ces mesures permettent de détecter l’existence d’un stress réplicatif lié à une diminution des pools de nucléotides ou une inhibition des DNA polymérases (FV réduite), à des dommages à l’ADN (FA élevée), une diminution locale ou générale du licensing ou de l’activation des origines (IOD élevée, GIFD réduite)(Bialic et al., 2015).
Que pouvons-nous vous apporter ?
Plusieurs prestations sont possibles :
• La production et la distribution de lamelles silanisées pour les personnes souhaitant réaliser eux-mêmes leurs expériences de peignage moléculaire (service limité aux laboratoires CNRS ou affiliés CNRS, pour cause de propriété intellectuelle).
• L’analyse biologique complète pour toute personne désirant nous confier ses échantillons, du traitement des blocs d’agarose jusqu’à l’analyse des images de peignage et les analyses statistiques.
• L’analyse des images de peignage moléculaire : l’utilisateur nous envoie ses images et nous nous chargeons de mesurer les paramètres de la réplication de son choix (vitesse de fourches, densité globale, asymétries de fourches et distance inter-origine) via notre logiciel d’analyse propriétaire IDeFIx.
• Fabrication d’appareillage de peignage : Sur le modèle de notre machine existante (voir Figure 2).
• Enfin, de la formation est possible à tout personnel tout aussi bien sur les traitements biologiques que pour l’analyse d’images avec notre logiciel dédié.
Qui contacter ?
• Le responsable de la plateforme / responsable scientifique : Etienne Schwob etienne.schwob@igmm.cnrs.fr au 04.34.35.96.79
• La responsable technique : Marjorie Drac marjorie.drac@igmm.cnrs.fr au 04.34.35.96.31 ou au 04.34.35.96.77
Où nous trouver / nous écrire ?
MONTPELLIER DNA COMBING FACILITY (Plateforme MDC)
Institut Génétique Moléculaire de Montpellier (IGMM)
CNRS UMR5535
1919 route de Mende
34293 Montpellier Cedex 5
France
Références:
Bensimon, A., Simon, A., Chiffaudel, A., Croquette, V., Heslot, F., and Bensimon, D. (1994). Alignment and sensitive detection of DNA by a moving interface. Science 265, 2096–2098.
Bialic, M., Coulon, V., Drac, M., Gostan, T., and Schwob, E. (2015). Analyzing the dynamics of DNA replication in Mammalian cells using DNA combing. Methods Mol. Biol. Clifton NJ 1300, 67–78.
Lengronne, A., Pasero, P., Bensimon, A., and Schwob, E. (2001). Monitoring S phase progression globally and locally using BrdU incorporation in TK(+) yeast strains. Nucleic Acids Res. 29, 1433–1442.
Michalet, X., Ekong, R., Fougerousse, F., Rousseaux, S., Schurra, C., Hornigold, N., van Slegtenhorst, M., Wolfe, J., Povey, S., Beckmann, J.S., et al. (1997). Dynamic molecular combing: stretching the whole human genome for high-resolution studies. Science 277, 1518–1523.