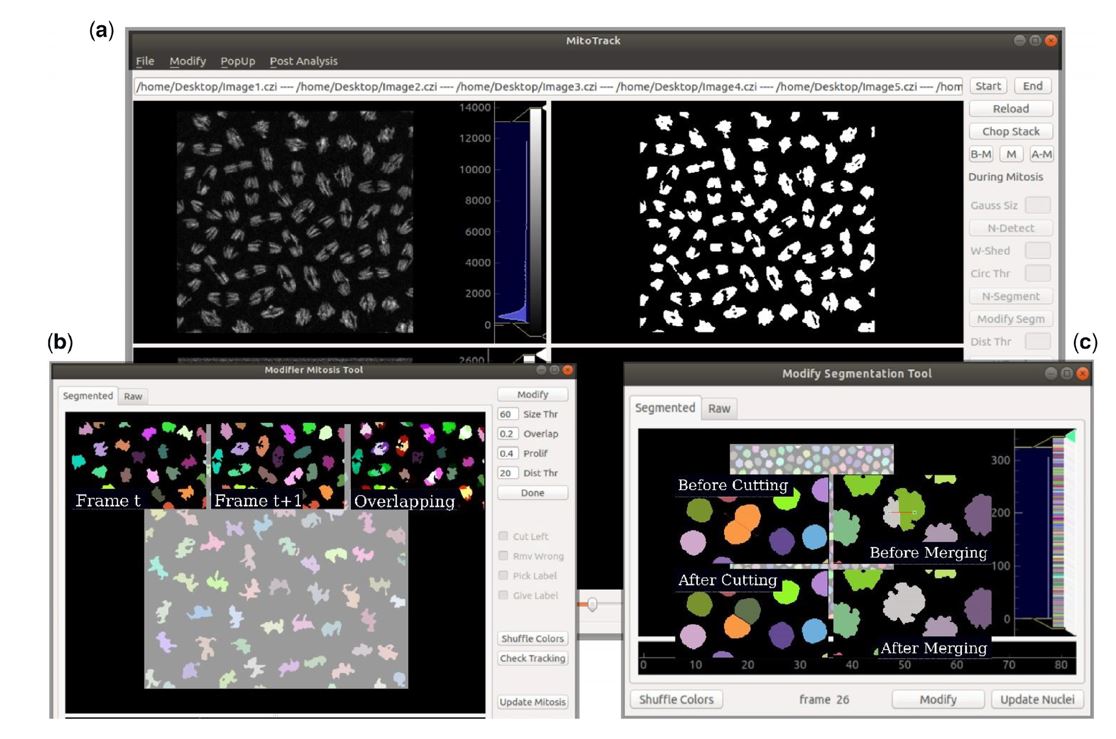
Au cours du développement, les cellules progénitrices subissent de multiples cycles de divisions cellulaires au cours desquels les programmes de transcription doivent être propagés fidèlement. Pour étudier la transmission mitotique de ces programmes, il est essentiel de pouvoir suivre les activités de transcription et leur transmission des cellules mères aux cellules filles dans des embryons vivants. Compte tenu de la nature stochastique de ce phénomène, il est nécessaire de collecter et d’analyser une grande quantité de données pour examiner la distribution statistique des résultats. À cette fin, nous avons développé MitoTrack, un logiciel développé en PythonTM avec une interface utilisateur graphique conviviale qui fournit des outils graphiques pour les corrections manuelles, la visualisation et le contrôle des résultats. Afin d’étudier l’activation temporelle de la transcription dans des cycles cellulaires consécutifs, MitoTrack suit les noyaux à travers la mitose en conservant les informations de lignage et en détectant l’apparition des sites de transcriptions. En conséquence, nous pouvons extraire le temps d’activation de chaque noyau et de ses filles, ce qui nous permet d’étudier la mémoire de transcription à travers la mitose.
Bioinformatics. 2019 Oct 3. pii: btz717. doi: 10.1093/bioinformatics/btz717. [Epub ahead of print]
MitoTrack, a user-friendly semi-automatic software for lineage tracking in living embryos.
Trullo A, Dufourt J, Lagha M.
Unless provided in the caption above, the following copyright applies to the content of this slide: © The Author(s) 2019. Published by Oxford University Press.This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0/), which permits non-commercial re-use, distribution, and reproduction in any medium, provided the original work is properly cited. For commercial re-use, please contact journals.permissions@oup.com
Bioinformatics, btz717, https://doi.org/10.1093/bioinformatics/btz717
The content of this slide may be subject to copyright: please see the slide notes for details.