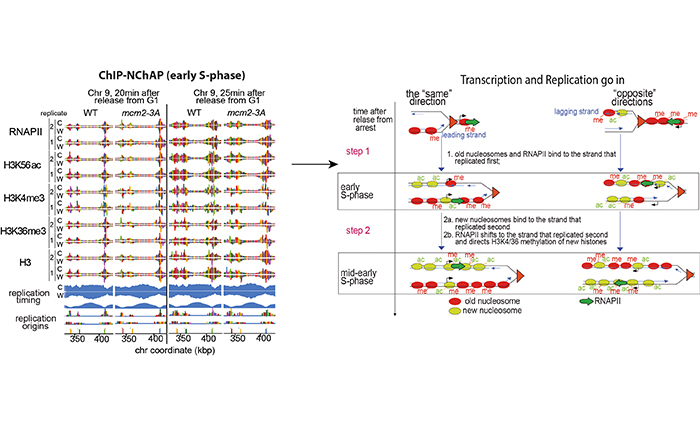
The asymmetric distribution of RNA polymerase II and nucleosomes on replicated daughter genomes is caused by differences in replication timing between the lagging and the leading strand
26 janvier 2022
Nous avons développé ChIP-NChAP (Nascent Chromatin Avidin Pulldown), une méthode permettant de suivre la réplication, la transcription et la dynamique de maturation de la structure chromatinienne sur chaque génome fille. Nous trouvons une différence dans le timing de réplication des brins tardifs par rapport aux brins précoces de l’ordre de quelques minutes dans la plupart des gènes de levure. Nous proposons un modèle dans lequel la majorité des anciennes histones et RNAPII se lient à la copie du gène qui s’est répliquée en premier, tandis que les nucléosomes nouvellement synthétisés sont assemblés sur la copie qui s’est répliquée en second. L’enrichissement en RNAPII bascule ensuite à la copie sœur qui a été répliquée en second. L’ordre de réplication est largement déterminé par l’orientation génique : si la transcription et la réplication avancent dans le même sens, le brin précoce se réplique en premier ; si elles vont dans le sens opposé, le brin tardif se réplique en premier. Nous proposons que les anciennes histones caractéristiques des gènes activement transcrits sont héritées simultanément et indépendamment des complexes de transcription. La transcription contribue ainsi activement au rétablissement de l’état actif de la chromatine.