Toutes les cellules vivantes s'appuient sur l'établissement de programmes d'expression génique particuliers pour assurer les fonctions cellulaires de base, acquérir des identités spécifiques et répondre à divers stimuli extracellulaires et intracellulaires. La première étape de l'expression génétique, et probablement la plus régulée, est la transcription de l'ADN génomique par les ARN polymérases, qui génèrent des répertoires distincts de transcrits d'ARN (transcriptomes) en fonction du type de cellule et des conditions environnementales. Cependant, même si la transcription est absolument essentielle, elle est aussi une source de conflits potentiels avec d'autres processus coexistant dans le génome, comme la réplication de l'ADN et la réparation des lésions de l'ADN. S'ils ne sont pas résolus, ces conflits peuvent mettre en péril l'intégrité du génome.
Nous cherchons à élucider les voies qui déterminent la composition des transcriptomes et les mécanismes qui régulent les conflits causés par les machineries de transcription. Nous cherchons également à comprendre comment les perturbations de ces processus sont impliquées dans des maladies humaines. Nous utilisons une variété d'approches allant de la génomique à haute résolution à la génétique moléculaire et la biochimie et nous utilisons différents systèmes modèles eucaryotes tels que la levure bourgeonnante et divers types de cellules humaines, y compris les motoneurones.
Notre recherche est structurée en 4 axes différents :
AXE 1:
Étude du mécanisme de terminaison de la transcription des gènes non codants et codant pour les protéines.
(PIs: D. Libri & O. Porrua)
La transcription n'est pas limitée aux régions codant pour des ARN fonctionnels connus, mais a lieu virtuellement partout dans le génome. Ce phénomène est appelé transcription pervasive ou cachée (voir nos revues Villa & Porrua, 2022 ; et Jensen et al, 2013) et est conservé dans tous les organismes de la bactérie à l'homme.
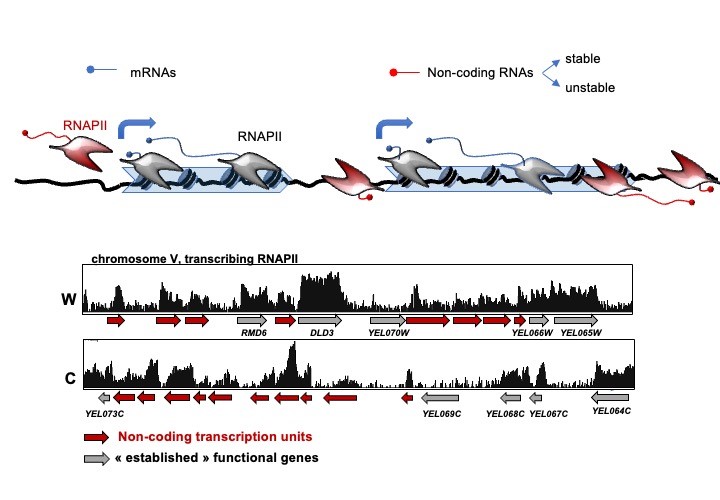
L’étendue des événements de transcription non-codants doit être contrôlée pour éviter des interférences avec l'expression des gènes voisins et d'autres processus associés à l'ADN. La terminaison de la transcription joue un rôle important dans ce contexte (voir notre revue : Porrua & Libri, 2015), et l'étude des mécanismes impliqués dans ce processus est l'un de nos principaux intérêts. La terminaison de la transcription à l'extrémité 3' des gènes codant pour des protéines dépend d'un complexe multi-sous-unités conservé appelé le complexe CPF-CF. Chez la levure bourgeonnante, le complexe NNS, composé des protéines de liaison à l'ARN Nrd1 et Nab3, et de l'hélicase Sen1, termine les événements de transcription pervasive mais aussi la transcription de gènes d'ARN non-codants fonctionnels tels que les snoRNAs. De plus, le complexe NNS favorise la dégradation des ARN non-codants par l'exosome nucléaire et son cofacteur le complexe TRAMP (figure 2).
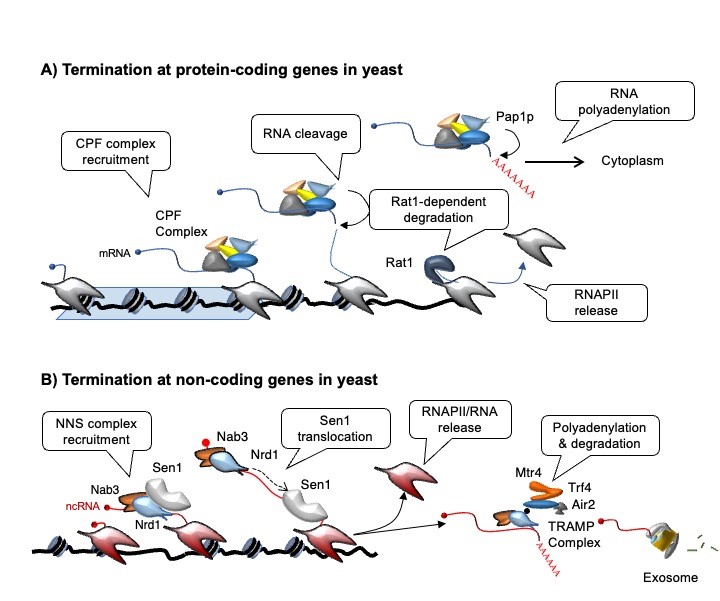
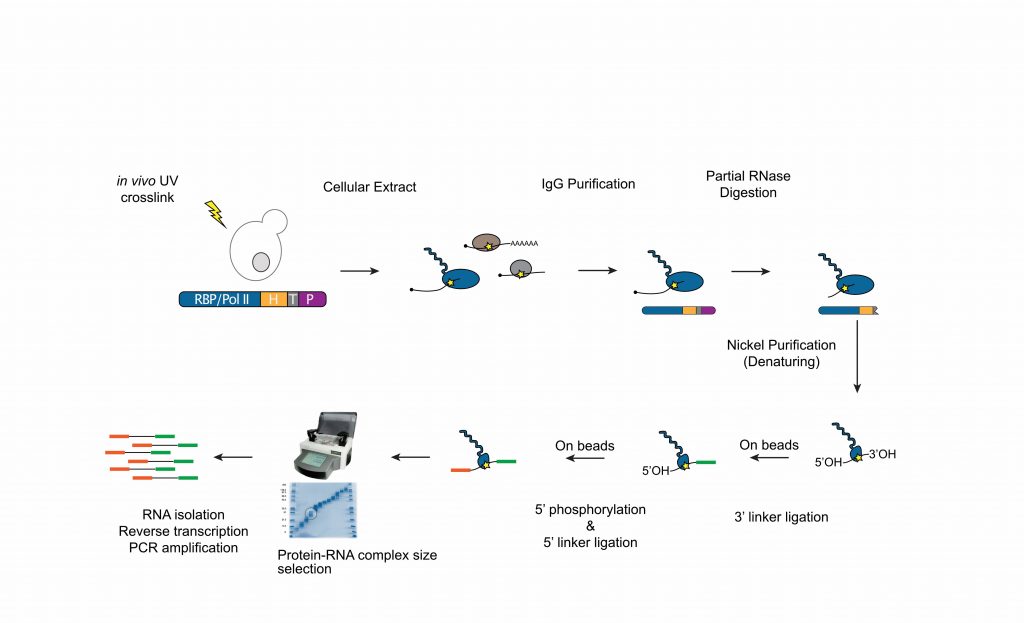
Au cours des dernières années, nous avons largement caractérisé la fonction du complexe NNS. Cependant, de nombreux aspects des mécanismes de terminaison restent obscurs, aussi bien pour les gènes non-codants que pour les gènes codant pour ARNm. Nous utilisons une technique qui permet de détecter in vivo la position de l’ARNpol II avec une résolution d'un seul nucléotide (CRAC, Crosslinking Analysis of cDNAs, figure 3, Bohnsack et al., 2012 ; Challal et al., 2022) pour générer des cartes de transcription dans des mutants du complexe NNS et de la voie CPF-CF.
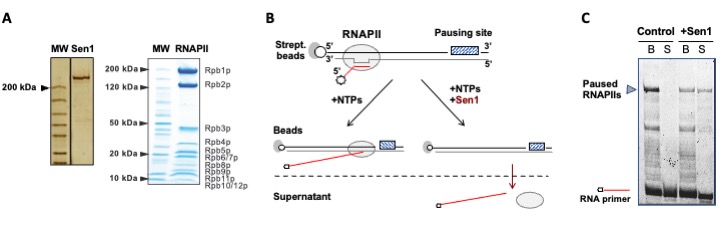
Cela permet de réaliser des analyses comparatives des différentes voies de terminaison et fournit des informations sur les mécanismes (ainsi que des résultats inattendus !). Nous aimons combiner ces analyses à haute résolution à l'échelle du génome avec des approches biochimiques in vitro (figure 4) et structurelles pour étudier les mécanismes de terminaison de la transcription et l'interaction entre les différentes voies de terminaison (comme exemple typique, voir notre publication récente : Xie et al, 2022).
AXE 2:
Analyse de l'impact de la transcription non-codante dans l'expression des gènes dans différentes conditions physiologiques
(PI : D. Libri & O. Porrua)
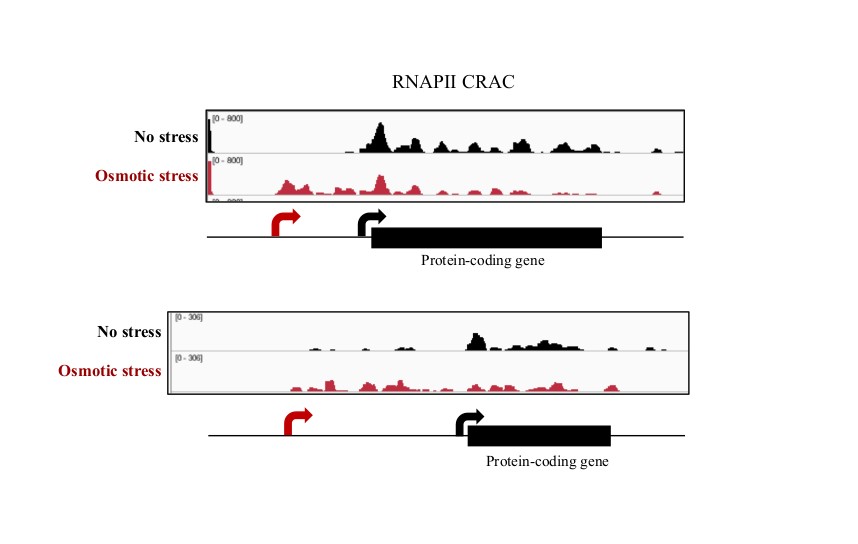
Les événements de transcription non-codante peuvent réguler l'expression des gènes en affectant la fonction des promoteurs des gènes voisins. Le potentiel régulateur de la transcription non-codante a été généralement négligé, principalement parce que de nombreuses analyses antérieures reposaient sur la détection de l'ARN comme proxy de la transcription et que les ARNnc produits par des événements de transcription potentiellement régulateurs sont souvent instables et difficilement détectables dans les cellules sauvages. La cartographie à haute résolution et directionnelle de l’ARNpol II en cours de transcription permet de contourner ce problème.
Nous sommes intéressés à explorer davantage les voies de régulation par la transcription non-codante dans différentes conditions physiologiques et de stress. Nous avons détecté de nombreux nouveaux événements de transcription d'ARNnc dans ces conditions, dont certains dérivent de promoteurs bidirectionnels activés, d'autres d'unités de transcription non-codantes "solitaires" et d'autres encore d'une efficacité réduite de la terminaison de la transcription (voir notre récente publication Haidara et al, 2022 ; et figure 5). Nous poursuivons l'étude de l'impact de la transcription non-codante sur l'expression des gènes en utilisant une variété d'approches et d'outils bioinformatiques.
AXE 3 :
Caractérisation des mécanismes responsables de la résolution des conflits transcription-réplication
(PI : D. Libri)
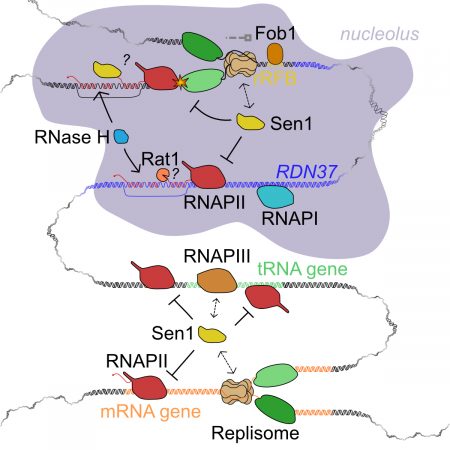
L'existence d'événements de transcription qui transcendent les limites des gènes canoniques annotés est un défi majeur pour la cohabitation de la transcription et d'autres événements associés à l'ADN comme la réplication. Nous nous sommes intéressés dans un passé récent à l'impact que la transcription pervasive a sur la fonction des origines de réplication de la levure (voir Candelli et al, 2018). Nous orientons maintenant nos intérêts vers les conflits générés par la transcription par l’ARNpol II. Dans une série d'études récentes (Aiello et al., 2022 ; Appanah et al., 2020), nous avons analysé les relations entre la transcription par l’ARNpol II et les événements de réplication et de transcription médiés par d'autres polymérases. Nous avons démontré, en collaboration avec les laboratoires de Piccoli, Pasero et Palancade, que Sen1, indépendamment de son rôle dans la terminaison de la transcription non-codante, a aussi un rôle capital dans le contrôle des conflits transcription-réplication et transcription-transcription. Nous avons montré que Sen1 élimine les ARNpols II qui entrent en collision avec le réplisome ou avec d'autres ARN polymérases en cours de transcription, se qualifiant ainsi comme un régulateur majeur de l'encombrement génomique (Figure 6).
Lorsque des conflits se produisent, l'ARN naissant peut s'hybrider au brin d'ADN matrice, formant des structures appelées R-loops. Ces structures peuvent générer des dommages à l'ADN et sont généralement éliminées par les RNases H qui dégradent la partie ARN de l'hybride. On pense également que Sen1 joue un rôle dans la limitation de la formation des R-loops ou dans leur résolution une fois qu'ils sont formés. Nous avons étudié le rôle des R-loops et des RNases H dans les régions de conflits, et décrit une nouvelle méthode de détection des R-loops à haute résolution. Nous poursuivrons les études sur le mécanisme de résolution des conflits, les rôles des RNases H et des R-loops dans ces processus et l'impact sur la stabilité du génome.
AXIS 4 :
Étude de la fonction moléculaire de la sénataxine humaine et de son implication dans les maladies neurodégénératives
(PI : O.Porrua)
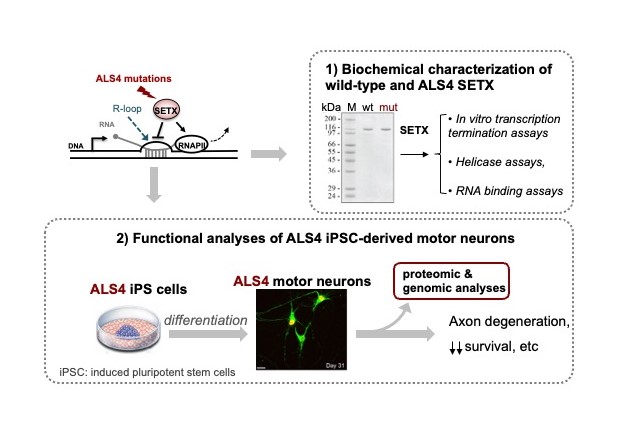
L'homologue humain de Sen1, la sénataxine (SETX), a attiré beaucoup d'attention en raison de son lien avec deux maladies neurodégénératives. Des mutations récessives de perte de fonction de SETX ont été associées à l'ataxie avec apraxie oculomotrice de type 2 (AOA2), tandis que des mutations dominantes de gain de fonction de SETX sont liées à une forme juvénile de sclérose latérale amyotrophique (SLA) appelée SLA4 (ALS4 en anglais). Comme Sen1, SETX a été attribuée un rôle dans la terminaison de la transcription ainsi que dans la résolution des R-loops, cependant le rôle précis de SETX dans ces processus restait mal compris à cause de l'absence de données biochimiques sur les propriétés et les activités de SETX. De plus, une identification systématique des protéines et ARNs interagissant avec SETX n’a pas été réalisée. Pour combler ces lacunes, nous avons mis à profit notre expertise et les outils que nous avons développés pour la caractérisation fonctionnelle de l'homologue de SETX chez la levure pour élucider la fonction moléculaire de SETX. En collaboration avec M. Sebesta et R. Stefl, nous avons récemment purifié le domaine catalytique de SETX et, en utilisant une variété d’essais biochimiques in vitro, nous avons montré pour la première fois que SETX est une hélicase capable de résoudre des R-loops et un facteur de terminaison de la transcription (voir notre pre-print Hasanova et al, 2022, https://www.biorxiv.org/content/10.1101/2022.08.25.505353v1).
De plus, nous étudions actuellement les bases moléculaires de la SLA associée à SETX en collaboration avec S. Nedelec (IFM, Paris). A cette fin, nous avons généré des motoneurones humains à partir de cellules souches pluripotentes induites portant des mutations SLA4 et nous utilisons une variété d'approches pour étudier l'impact de ces mutations sur la physiologie des motoneurones. Ensuite, nous combinerons des approches biochimiques, protéomiques et génomiques pour dévoiler les dérégulations responsables de la dégénérescence des motoneurones dans la SLA4 (figure 7).
Recent Publications :
1- Aiello, U., Challal D., Wentzinger, G., Lengronne, A., Appanah, R., Pasero, P., Palancade, B. and Libri, D*. (2022). Sen1 is a master regulator of transcription-driven conflicts. Cell S1097-2765(22)00604-9. PMID: 35839782
This work was highlighted by the CNRS
2- Xie, J., Aiello, U., Clement, Y., Haidara, N., Girbig, M., Schmitzova, J., Pena, P. Müller, C.W., Libri, * and Porrua, O*. An integrated model for termination of RNA polymerase III transcription. Sci Adv. 8(28):eabm9875. PMID: 35857496
This work was highlighted by the CNRS
3- Han, Z., Jasnovidova, O., Haidara, N., Tudek, A., Kubicek, K., Libri, D., Stefl, R. and Porrua, O* (2020). Termination of non-coding transcription in yeast relies on both an RNA Pol II CTD-interaction domain and a CTD-mimicking region in Sen1. EMBO J 39(7):e101548. PMID: 32107786
This work was recommended in Faculty Opinions.
4- Challal, D., Barucco, M., Kubik, S., Feuerbach, F., Candelli, T., Geoffroy, H., Benaksas, C., Shore, D., and Libri, D*. (2018). General Regulatory Factors Control the Fidelity of Transcription by Restricting Non-coding and Ectopic Initiation. Cell 72, 955-969.e7. PMID: 30576657
List of all the publication :
*co-corresponding authors
Reviews in italic
Hasanova, Z., Klapstova, V., Porrua*, O., Stefl*, R., and Sebesta*, M. (2022). Human senataxin is a bona fide R-loop resolving enzyme and transcription termination factor. 2022.08.25.505353. https://doi.org/10.1101/2022.08.25.505353.
Girbig, M., Xie, J., Grötsch, H., Libri, D., Porrua, O., and Müller, C.W. (2022). Architecture of the yeast Pol III pre-termination complex and pausing mechanism on poly(dT) termination signals. Cell Reports 40. https://doi.org/10.1016/j.celrep.2022.111316.
Villa, T. and Porrua, O (2022). Pervasive transcription: a controlled risk. FEBS J. doi: 10.1111/febs.16530. PMID: 35587776
Haidara, N., Giannini, M. and Porrua, O (2022). Modulated termination of non-coding transcription partakes in the regulation of gene expression. Nucleic Acids Res 50(3):1430-1448. PMID: 35037029.
Challal, D., Colin, J., Villa, T., and Libri, D. (2022). A Modified Cross-Linking Analysis of cDNAs (CRAC ) Protocol for Detecting RNA-Protein Interactions and Transcription at Single-Nucleotide Resolution. Methods Mol Biol 2477, 35–55. https://doi.org/10.1007/978-1-0716-2257-5_3.
Scutenaire, J., Plassard, D., Matelot, M., Villa, T., Zumsteg, J., Libri, D., and Séraphin, B. (2022). The S. cerevisiae m6A-reader Pho92 promotes timely meiotic recombination by controlling key methylated transcripts. Nucleic Acids Res gkac640. https://doi.org/10.1093/nar/gkac640.
Aiello, U., Challal, D., Wentzinger, G., Lengronne, A., Appanah, R., Pasero, P., Palancade, B., and Libri, D. (2022). Sen1 is a key regulator of transcription-driven conflicts. Mol Cell 82, 2952-2966.e6. https://doi.org/10.1016/j.molcel.2022.06.021.
Xie, J., Aiello, U., Clement, Y., Haidara, N., Girbig, M., Schmitzova, J., Pena, V., Müller, C.W., Libri, D., and Porrua, O. (2022). An integrated model for termination of RNA polymerase III transcription. Sci Adv 8, eabm9875. https://doi.org/10.1126/sciadv.abm9875.
Villa, T., Barucco, M., Martin-Niclos, M.-J., Jacquier, A., and Libri, D. (2020). Degradation of Non-coding RNAs Promotes Recycling of Termination Factors at Sites of Transcription. Cell Rep 32, 107942.
Porrua, O. (2020). Purification and in vitro analysis of the exosome cofactors Nrd1-Nab3 and Trf4-Air2. Methods Mol Biol 2062:277-289. PMID: 31768982
Begley, V., Jordán-Pla, A., Peñate, X., Garrido-Godino, A.I., Challal, D., Cuevas-Bermúdez, A., Mitjavila, A., Baruc co, M., Gutiérrez, G., Singh, A., Alepuz, P., Navarro, F., Libri* , D., Perez-Ortin*, JE., Chavez*, D. (2020). Xrn1 influence on gene transcription results from the combination of general effects on elongating RNA pol II and gene-specific chromatin configuration. RNA Biol 1–14.
Appanah, R., Lones, E.C., Aiello, U., Libri, D., and De Piccoli, G. (2020). Sen1 Is Recruited to Replication Forks via Ctf4 and Mrc1 and Promotes Genome Stability. Cell Rep 30, 2094-2105.e9.
Han, Z., Jasnovidova, O., Haidara, N., Tudek, A., Kubicek, K., Libri, D., Stefl, R., and Porrua, O. (2020). Termination of non-coding transcription in yeast relies on both an RNA Pol II CTD interaction domain and a CTD-mimicking region in Sen1. EMBO J. e101548.
Kubik, S., Bruzzone, M.J., Challal, D., Dreos, R., Mattarocci, S., Bucher, P., Libri, D., and Shore, D. (2019). Opposing chromatin remodelers control transcription initiation frequency and start site selection. Nat. Struct. Mol. Biol. 26, 744–754.
Wang, S., Han, Z., Libri, D., Porrua, O., and Strick, T.R. (2019). Single-molecule characterization of extrinsic transcription termination by Sen1 helicase. Nat Commun 10, 1545.
Han, Z. and Porrua, O. (2018). Helicases as transcription termination factors: Different solutions for a common problem. Transcription. 9(3):152-158. PMID: 28886303
Candelli, T., Gros*, J., and Libri*, D. (2018). Pervasive transcription fine-tunes replication origin activity. Elife 7.
Challal, D., Barucco, M., Kubik, S., Feuerbach, F., Candelli, T., Geoffroy, H., Benaksas, C., Shore, D., and Libri, D. (2018). General Regulatory Factors Control the Fidelity of Transcription by Restricting Non-coding and Ectopic Initiation. Mol. Cell 72, 955-969.e7.
Candelli, T., Challal, D., Briand, J.-B., Boulay, J., Porrua, O., Colin, J., and Libri, D. (2018). High-resolution transcription maps reveal the widespread impact of roadblock termination in yeast. EMBO J. 37.
Luciano, P.°, El-kaoutari°, A., Challal° D., Jeon J., Candelli, T., Jourquin, F., Kim, J.*, Libri, D.*, and Geli, V.* (2017) Binding to RNA in vivo regulates Set1 function, Cell Discov 3, 17040.
Simonetti, F., Candelli,T., Leon, S., Libri, D., Rougemaille, M. (2017) Ubiquitination-dependent control of sexual differentiation in fission yeast. Elife 6
Leonaitė, B., Han, Z., Basquin, J., Bonneau, F., Libri, D., Porrua*, O., and Conti*, E. (2017). Sen1 has unique structural features grafted on the architecture of the Upf1-like helicase family. EMBO J.
Han, Z., Libri, D., and Porrua, O. (2017). Biochemical characterization of the helicase Sen1 provides new insights into the mechanisms of non-coding transcription termination. Nucleic Acids Res. 45, 1355–1370.
Babour, A., Shen, Q., Dos-Santos, J., Murray, S., Gay, A., Challal, D., Fasken, M., Palancade, B., Corbett, A., Libri, D., Mellor, J. and Dargemont, C. (2016). The Chromatin Remodeler ISW1 Is a Quality Control Factor that Surveys Nuclear mRNP Biogenesis. Cell 167, 1201–1214.e15.
Porrua, O., Boudvillain, M., and Libri, D. (2016). Transcription Termination: Variations on Common Themes. Trends Genet. 32, 508–522.
Libri, D. (2015). Sleeping Beauty and the Beast (of pervasive transcription). RNA 21, 678–679.
Porrua, O., and Libri, D. (2015a). Transcription termination and the control of the transcriptome: why, where and how to stop. Nat. Rev. Mol. Cell Biol. 16, 190–202.
Porrua, O., and Libri, D. (2015b). Characterization of the mechanisms of transcription termination by the helicase Sen1. Methods Mol. Biol. 1259, 313–331.
Tudek, A., Candelli, T., and Libri, D. (2015). Non-coding transcription by RNA polymerase II in yeast: Hasard or nécessité? Biochimie. 117, 28-36
Zhang, W., Collinet, B., Graille, M., Daugeron, M.-C., Lazar, N., Libri, D., Durand, D., and van Tilbeurgh, H. (2015). Crystal structures of the Gon7/Pcc1 and Bud32/Cgi121 complexes provide a model for the complete yeast KEOPS complex. Nucleic Acids Res. 43, 3358–3372.
Libri, D. (2015). Endless Quarrels at the End of Genes. Mol. Cell 60, 192–194.
Colin, J., Candelli, T., Porrua, O., Boulay, J., Zhu, C., Lacroute, F., Steinmetz, L.M., and Libri, D. (2014). Roadblock termination by reb1p restricts cryptic and readthrough transcription. Mol. Cell 56, 667–680.
Tudek*, A., Porrua*, O., Kabzinski, T., Lidschreiber, M., Kubicek, K., Fortova, A., Lacroute, F., Vanacova, S., Cramer, P., Stefl, R. and Libri, D. (2014). Molecular basis for the dual role of Nrd1p CID in transcription termination and non-coding RNA degradation. Mol. Cell, 55, 467-81
Mouaikel, J., Causse, S.Z., Rougemaille, M., Daubenton-Carafa, Y., Blugeon, C., Lemoine, S., Devaux, F., Darzacq, X., and Libri, D. (2013). High-Frequency Promoter Firing Links THO Complex Function to Heavy Chromatin Formation. Cell Rep 5, 1082-1094.
Lenstra*, T.L., Tudek*, A., Clauder, S., Xu, Z., Pachis, S.T., van Leenen, D., Kemmeren, P., Steinmetz, L.M., Libri*, D., and Holstege*, F.C. (2013). The Role of Ctk1 Kinase in Termination of Small Non-Coding RNAs. PLoS One 8, e80495.
Jensen, T.H., Jacquier, A., and Libri, D. (2013). Dealing with pervasive transcription. Mol Cell 52, 473-484.
Porrua, O., and Libri, D. (2013). A bacterial-like mechanism for transcription termination by the Sen1p helicase in budding yeast. Nat Struct Mol Biol 20, 884-891.
Porrua, O., and Libri, D. (2013). RNA quality control in the nucleus: the Angels’ share of RNA. Biochim. Biophys. Acta 1829, 604–611.
Rougemaille, M., Braun, S., Coyle, S., Dumesic, P.A., Garcia, J.F., Isaac, R.S., Libri, D., Narlikar, G.J., and Madhani, H.D. (2012). Ers1 links HP1 to RNAi. Proc. Natl. Acad. Sci. U.S.A. 109, 11258–11263.
Porrua, O., Hobor, F., Boulay, J., Kubicek, K., D'Aubenton-Carafa, Y., Gudipati, R.K., Stefl, R., and Libri, D. (2012). In vivo SELEX reveals novel sequence and structural determinants of Nrd1-Nab3-Sen1-dependent transcription termination. Embo J 31, 3935-3948.
Gudipati, R.K., Xu, Z., Lebreton, A., Seraphin, B., Steinmetz, L.M., Jacquier, A., and Libri, D. (2012). Extensive degradation of RNA precursors by the exosome in wild-type cells. Mol Cell 48, 409-421.
Rougemaille, M., and Libri, D. (2011). Control of cryptic transcription in eukaryotes. Adv Exp Med Biol 702, 122-131.
Daugeron, M.C., Lenstra, T.L., Frizzarin, M., El Yacoubi, B., Liu, X., Baudin-Baillieu, A., Lijnzaad, P., Decourty, L., Saveanu, C., Jacquier, A., Holstege, F.C.P., de Crécy-Lagard, V., van Tilbeurgh, H. and Libri, D. (2011). Gcn4 misregulation reveals a direct role for the evolutionary conserved EKC/KEOPS in the t6A modification of tRNAs. Nucleic Acids Res 39, 6148-6160.
Colin, J., Libri, D., and Porrua, O. (2011). Cryptic transcription and early termination in the control of gene expression. Genet Res Int 2011, 653494.
Assenholt, J., Mouaikel, J., Saguez, C., Rougemaille, M., Libri, D., and Jensen, T.H. (2011). Implication of Ccr4-Not complex function in mRNA quality control in Saccharomyces cerevisiae. Rna 17, 1788-1794.
Libri, D. (2010). Nuclear poly(a)-binding proteins and nuclear degradation: take the mRNA and run? Mol Cell 37, 3-5.
Honorine, R., Mosrin-Huaman, C., Hervouet-Coste, N., Libri, D., and Rahmouni, A.R. (2010). Nuclear mRNA quality control in yeast is mediated by Nrd1 co-transcriptional recruitment, as revealed by the targeting of Rho-induced aberrant transcripts. Nucleic Acids Res 39, 2809-2820.
Colin, J., Libri, D., and Villa, T. (2010). Sex matters in the birth of genes. Cell Res 20, 499-501.
Villa, T., Rougemaille, M., and Libri, D. (2008). Nuclear quality control of RNA polymerase II ribonucleoproteins in yeast: Tilting the balance to shape the transcriptome. Biochim Biophys Acta 1779, 524-531.
Thiebaut, M., Colin, J., Neil, H., Jacquier, A., Seraphin, B., Lacroute, F., and Libri, D. (2008). Futile cycle of transcription initiation and termination modulates the response to nucleotide shortage in S. cerevisiae. Mol Cell 31, 671-682.
Schmid, M., Rougemaille, M., Libri, D., and Jensen, T.H. (2008). Chapter 10. Estimating nuclear mRNA decay in Saccharomyces cerevisiae. Methods Enzymol 449, 205-219.
Rougemaille, M., Villa, T., Gudipati, R.K., and Libri, D. (2008). mRNA journey to the cytoplasm: attire required. Biol Cell 100, 327-342.
Rougemaille, M., Dieppois, G., Kisseleva-Romanova, E., Gudipati, R.K., Lemoine, S., Blugeon, C., Boulay, J., Jensen, T.H., Stutz, F., Devaux, F., and Libri, D. (2008). THO/Sub2p functions to coordinate 3'-end processing with gene-nuclear pore association. Cell 135, 308-321.
Libri, D. (2008). Quality control: Cell and the city. Biochim Biophys Acta 1779, 523.
Hecker, A., Lopreiato, R., Graille, M., Collinet, B., Forterre, P., Libri, D., and van Tilbeurgh, H. (2008). Structure of the archaeal Kae1/Bud32 fusion protein MJ1130: a model for the eukaryotic EKC/KEOPS subcomplex. Embo J 27, 2340-2351.
Gudipati, R.K., Villa, T., Boulay, J., and Libri, D. (2008). Phosphorylation of the RNA polymerase II C-terminal domain dictates transcription termination choice. Nat Struct Mol Biol 15, 786-794.
Assenholt, J., Mouaikel, J., Andersen, K.R., Brodersen, D.E., Libri*, D., and Jensen*, T.H. (2008). Exonucleolysis is required for nuclear mRNA quality control in yeast THO mutants. Rna 14, 2305-2313.
Rougemaille, M., Gudipati, R.K., Olesen, J.R., Thomsen, R., Seraphin, B., Libri*, D., and Jensen*, T.H. (2007). Dissecting mechanisms of nuclear mRNA surveillance in THO/sub2 complex mutants. Embo J 26, 2317-2326.
Chauveau, F., Aissouni, Y., Hamm, J., Boutin, H., Libri, D., Duconge, F., and Tavitian, B. (2007). Binding of an aptamer to the N-terminal fragment of VCAM-1. Bioorg Med Chem Lett 17, 6119-6122.
Thiebaut, M., Kisseleva-Romanova, E., Rougemaille, M., Boulay, J., and Libri, D. (2006). Transcription termination and nuclear degradation of cryptic unstable transcripts: a role for the nrd1-nab3 pathway in genome surveillance. Mol Cell 23, 853-864.
Pestourie, C., Cerchia, L., Gombert, K., Aissouni, Y., Boulay, J., De Franciscis, V., Libri, D., Tavitian, B., and Duconge, F. (2006). Comparison of different strategies to select aptamers against a transmembrane protein target. Oligonucleotides 16, 323-335.
Kisseleva-Romanova, E., Lopreiato, R., Baudin-Baillieu, A., Rousselle, J.C., Ilan, L., Hofmann, K., Namane, A., Mann, C., and Libri, D. (2006). Yeast homolog of a cancer-testis antigen defines a new transcription complex. Embo J 25, 3576-3585.
Cerchia, L., D'Alessio, A., Amabile, G., Duconge, F., Pestourie, C., Tavitian, B., Libri, D., and de Franciscis, V. (2006). An autocrine loop involving ret and glial cell-derived neurotrophic factor mediates retinoic acid-induced neuroblastoma cell differentiation. Mol Cancer Res 4, 481-488.
Wyers, F., Rougemaille, M., Badis, G., Rousselle, J.C., Dufour, M.E., Boulay, J., Regnault, B., Devaux, F., Namane, A., Seraphin*, B., Libri*, D. and Jacquier* A. (2005). Cryptic pol II transcripts are degraded by a nuclear quality control pathway involving a new poly(A) polymerase. Cell 121, 725-737. * co-corresponding authors.
Olesen, J.R., Libri, D., and Jensen, T.H. (2005). A link between transcription and mRNP quality in Saccharomyces cerevisiae. RNA Biol 2, 45-48.
Jensen, T.H., Boulay, J., Olesen, J.R., Colin, J., Weyler, M., and Libri, D. (2004). Modulation of transcription affects mRNP quality. Mol Cell 16, 235-244.
Thomsen, R., Libri, D., Boulay, J., Rosbash, M., and Jensen, T.H. (2003). Localization of nuclear retained mRNAs in Saccharomyces cerevisiae. Rna 9, 1049-1057.
Jensen, T.H., Dower, K., Libri, D., and Rosbash, M. (2003). Early formation of mRNP: license for export or quality control? Mol Cell 11, 1129-1138.
Cerchia, L., Libri, D., Carlomagno, M.S., and de Franciscis, V. (2003). The soluble ectodomain of RetC634Y inhibits both the wild-type and the constitutively active Ret. Biochem J 372, 897-903.
Libri, D., Duconge, F., Levy, L., and Vinauger, M. (2002). A role for the Psi-U mismatch in the recognition of the 5' splice site of yeast introns by the U1 small nuclear ribonucleoprotein particle. J Biol Chem 277, 18173-18181.
Libri*, D., Dower, K., Boulay, J., Thomsen, R., Rosbash, M., and Jensen*, T.H. (2002). Interactions between mRNA export commitment, 3'-end quality control, and nuclear degradation. Mol Cell Biol 22, 8254-8266.
Cerchia, L., Hamm, J., Libri, D., Tavitian, B., and de Franciscis, V. (2002). Nucleic acid aptamers in cancer medicine. FEBS Lett 528, 12-16.
Libri, D., Graziani, N., Saguez, C., and Boulay, J. (2001). Multiple roles for the yeast SUB2/yUAP56 gene in splicing. Genes Dev 15, 36-41.
Jensen, T.H., Boulay, J., Rosbash, M., and Libri, D. (2001). The DECD box putative ATPase Sub2p is an early mRNA export factor. Curr Biol 11, 1711-1715.



