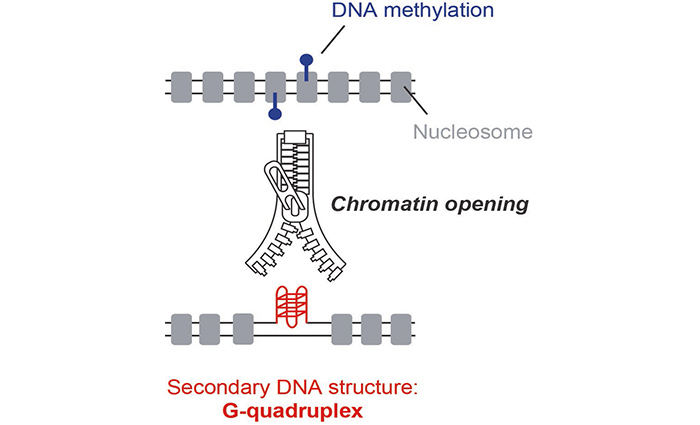
Dans ce travail, l’équipe de JC Andrau propose une nouvelle approche permettant d’identifier des structures secondaires de l’ADN, appelé G-quadruplexes (G4s) dans le contexte nucléaire. Cette technique, appelée G4access, utilise les propriétés d’une nuclease qui clive et digère l’ADN, dans des conditions où les G4s résistent à cette digestion et demeurent protégés de l’enzyme. Après séquençage des résidus de digestion, il est possible de déterminer la localisation des G4s dans le génome (Figure). Les données issues de ce travail ont montré que les G4s sont des déterminants essentiels des régions ouvertes du génome et qu’ils sont enrichis dans les régions active en transcription, bien que la transcription ne soit pas requise pour leur détection. Les G4s observés sont également enrichis sur les loci soumis à empreinte parentale et antagonistes à la méthylation de l’ADN. Finalement, la technique G4access a mis en lueur un mécanisme potentiel d’action d’un ligand de G4s.
Cette étude ouvre des perspectives importantes pour la compréhension de la contribution des structures secondaires de l’ADN dans l’expression, la duplication et la maintenance du génome. Elle offre également un cadre de lecture pour déterminer les cibles de ligands de G4s et leurs éventuelles améliorations à visée thérapeutique.
A voir aussi sur les actualités de l’INSB
Ces résultats sont publiés dans la revue Nature Genetics.
G4access identifies G-quadruplexes and their associations with open chromatin and imprinting control regions. Esnault C, Magat T, Zine El Aabidine A, Garcia-Oliver E, Cucchiarini A, Bouchouika S, Lleres D, Goerke L, Luo Y, Verga D, Lacroix L, Feil R, Spicuglia S, Mergny JL, Andrau JC. Nat Genet. 2023 Jul 3. doi: 10.1038/s41588-023-01437-4.
https://www.nature.com/articles/s41588-023-01437-4