Notre projet vise à comprendre les bases de la régulation de la transcription dans le contexte de la chromatine dans différents modèles en mettant l'accent sur le rôle des structures secondaires de l'ADN dans le contrôle de la transcription. Nous développons 4 thèmes principaux.
- Rôle des structures secondaires G-quadruplex de l'ADN dans l'activité du promoteur et d'autres modules de régulation par l'exclusion des nucléosomes
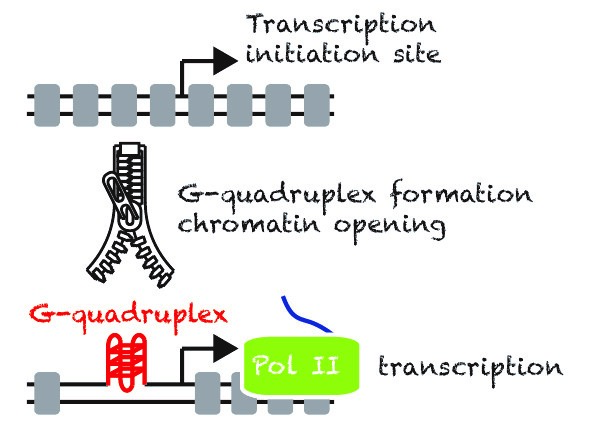
Malgré de nombreux efforts dans le passé, la communauté scientifique ne comprend toujours pas ce qui définit réellement un promoteur dans les cellules eucaryotes. Alors que la plupart des déterminants de séquence proposés pour les promoteurs sont peu conservés, nous proposons que les structures secondaires de l'ADN jouent un rôle essentiel dans le maintien de ces structures ouvertes à la chromatine par défaut. Notre modèle dans les cellules de mammifères promeut l'idée que les G-quadruplexes (G4s), qui sont des structures stables d'ADN simple brin, sont des modules d'exclusion des nucléosomes nécessaires mais non suffisants pour l'activité des promoteurs. Ainsi, au-delà des séquences primaires de l'ADN, ces structures peuvent jouer un rôle essentiel pour soutenir une activité promotrice robuste, plus spécifiquement au niveau des gènes hautement et largement exprimés.
Pour étayer nos hypothèses de travail, nous avons développé une nouvelle technique, G4access1, qui permet de repérer les séquences formant des G4s dans le contexte de la chromatine. En utilisant ces approches et d'autres, nos travaux récents ont montré que les G4s agissent comme des éléments promoteurs2 et sont mutuellement exclusifs avec la méthylation de l'ADN, y compris à des loci d'empreinte qui sont spécifiquement exprimés aux allèles maternels et paternels. Notre laboratoire cherche maintenant à mieux comprendre comment les G4s régulent non seulement les promoteurs, mais aussi les îlots CpG, les enhancers et les régions isolatrices. Nous étudions également comment les régulateurs G4 influencent l'expression des gènes, l'ouverture de la chromatine, l'enroulement et la stabilité de l'ADN.
Principales collaborations : Jean-Louis Mergny (Ecole Polytechnique, Orsay, France), Salvatore Spicuglia (TAGC, Marseille), Kateryna Makova (Penn state University), Dann Nooordemer (I2BC, Orsay).
- Bases moléculaires des chromatinopathies
Les chromatinopathies sont un groupe de maladies qui impliquent des mutations dans les protéines et les enzymes régulant la chromatine. Ces mutations sont le plus souvent acquises de novo et monogéniques. Certains complexes impliqués dans la méthylation ou l'acétylation des histones peuvent, par exemple, conduire aux syndromes de Kabuki ou de Rubinstein-Taybi. Ces syndromes peuvent entraîner des altérations neurologiques et notamment des anomalies visio-spatiales, des malformations cardiaques, des déficiences immunitaires ainsi que d'autres caractéristiques3-5. En étroite collaboration avec des cliniciens de l'hôpital de Montpellier et d'autres laboratoires européens, au sein d'un consortium européen MSCA Chrom_rare, nous développons des modèles humains d'iPS et de poisson zèbre pour étudier ces maladies, en nous concentrant sur le syndrome de Kabuki qui implique des mutations monogéniques des gènes KMT2D et KDM6A. Nous étudions également d'autres modèles haplo-insuffisants liés à Kabuki, tels que CDK13, afin de surveiller leurs altérations spécifiques de la chromatine et de l'expression génique, y compris, dans certains cas, l'altération de structures secondaires de l'ADN, telles que les G-quadruplexes. Avec nos collaborateurs, nous explorons également les possibilités de traitement liés à Kabuki dans des modèles animaux chez les poissons zèbres.
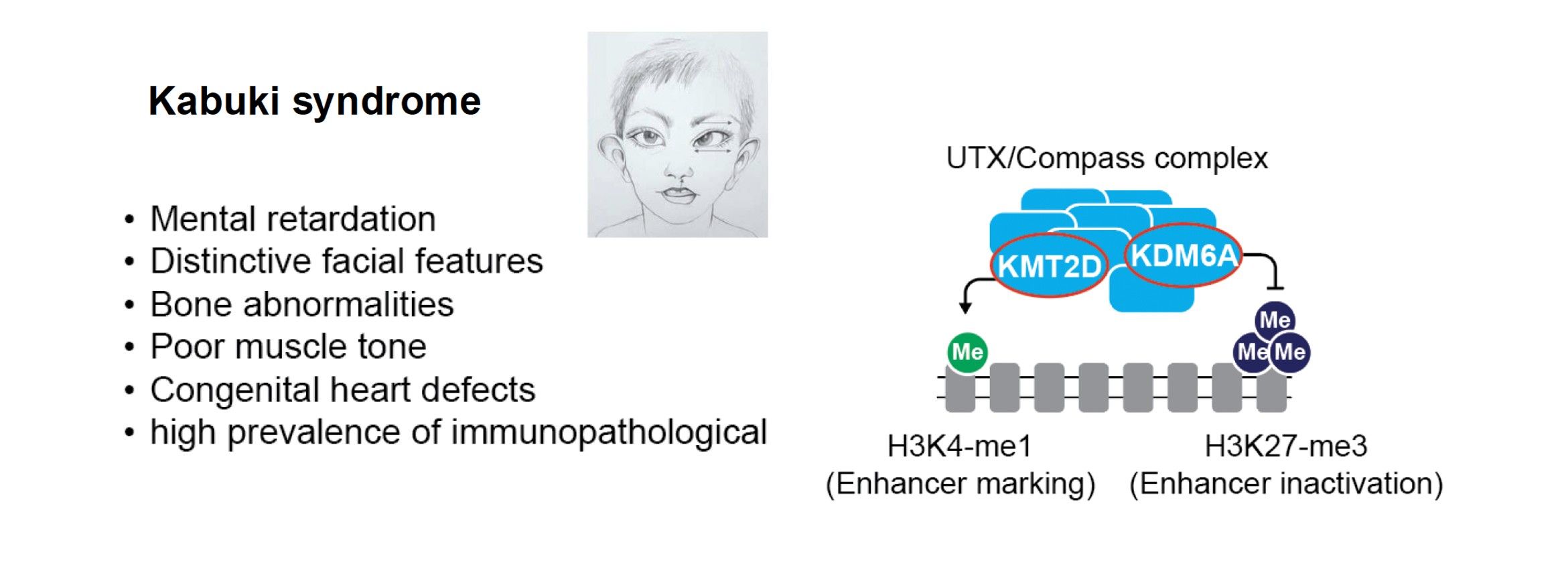
Principales collaborations : David Genevieve (CHU, Montpellier), Farida Djouad (IRMB, Montpellier), Aleksandra Pekowska (Nencki Institute, Warsaw, Poland), Siddharth Banka (Un. Manchester, UK).
- Rôle de la Mécano-transduction dans l’organisation 3D du génome et dans l’expression des gènes (programme dirigé par Cyril Esnault)
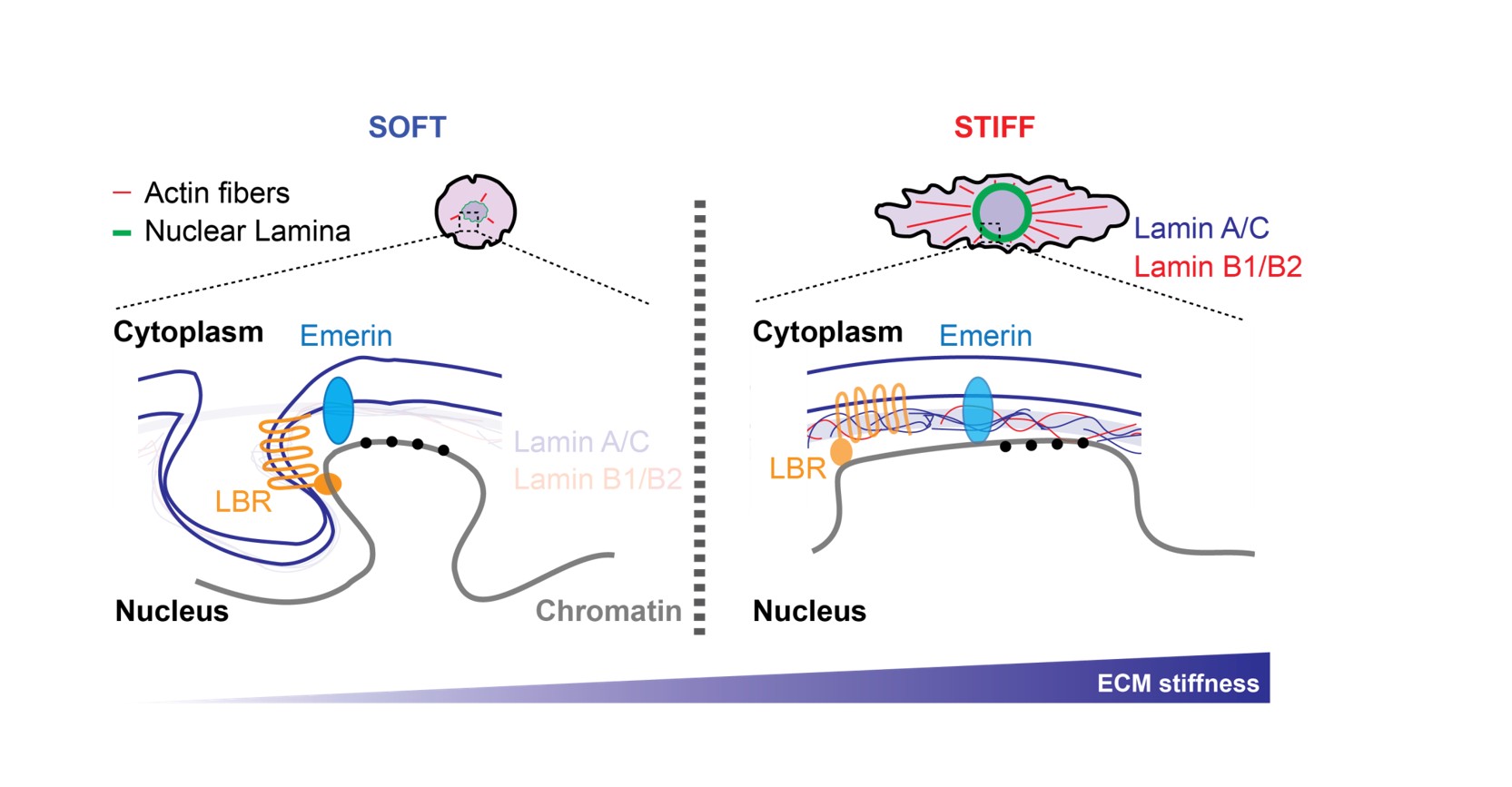
Les signaux environnementaux tels que ceux liés aux facteurs de croissance, aux hormones ou aux nutriments sont essentiels à la régulation du comportement cellulaire. Outre ces signaux biochimiques, les cellules réagissent également à des contraintes physiques, telles que l’étirement, la compression et la rigidité de la matrice extracellulaire (MEC). L'élasticité de la MEC, par exemple, revêt une importance physiologique cruciale en : (i) régulant les différenciations des cellules souches épithéliales et mésenchymateuses, (ii) favorisant la transformation des fibroblastes en fibroblastes associés au cancer (CAF) et (iii) favorisant la progression du cancer du sein. Ces processus s'appuient sur des changements dans la transcription et la programmation épigénétique. À l'aide d'approches omiques, nous étudions comment la dynamique de la membrane nucléaire et du cytosquelette façonne l'organisation 3D du génome et module la transcription génétique. Nos travaux les plus récents ont mis l'accent sur l'interaction entre la lamine et le complexe Polycomb PRC2 lors de la transition d'une matrice souple à une matrice rigide.
Principales collaborations : Kerstin Bistricky (CBI, Toulouse), Manoel Manghi (LPT, Toulouse).
- Régulation de la transcription
Dans le passé, notre laboratoire s'est intéressé au contrôle de la transcription par le CTD de l'ARN polymérase II, montrant l'importance de ce domaine et de ses résidus Tyr dans le contrôle de la terminaison6,7. Nous avons également montré que la nature dynamique des enhancers au cours de la différenciation peut conditionner la sélection des isoformes au cours de la différenciation des cellules T chez la souris8. Dans le cadre d'une collaboration, nous étudions maintenant comment la transcription se déroule dans l'état quiescent (G0), en utilisant la levure comme organisme modèle.
Principale collaboration : J. Wu, ICGB, Bordeaux
Références
1 Esnault, C. et al. G4access identifies G-quadruplexes and their associations with open chromatin and imprinting control regions. Nat Genet, doi:10.1038/s41588-023-01437-4 (2023).
2 Esnault, C. et al. G-quadruplexes are promoter elements controlling nucleosome exclusion and RNA polymerase II pausing. Nat Genet 57, 1981-1993, doi:10.1038/s41588-025-02263-6 (2025).
3 Aref-Eshghi, E. et al. Evaluation of DNA Methylation Episignatures for Diagnosis and Phenotype Correlations in 42 Mendelian Neurodevelopmental Disorders. Am J Hum Genet 106, 356-370, doi:10.1016/j.ajhg.2020.01.019 (2020).
4 Comel, M. et al. Abnormal Immune Profile in Individuals with Kabuki Syndrome. J Clin Immunol 45, 7, doi:10.1007/s10875-024-01796-5 (2024).
5 Margot, H. et al. Immunopathological manifestations in Kabuki syndrome: a registry study of 177 individuals. Genet Med 22, 181-188, doi:10.1038/s41436-019-0623-x (2020).
6 Shah, N. et al. Tyrosine-1 of RNA Polymerase II CTD Controls Global Termination of Gene Transcription in Mammals. Mol Cell 69, 48-61 e46, doi:10.1016/j.molcel.2017.12.009 (2018).
7 Yahia, Y. et al. RNA polymerase II CTD is dispensable for transcription and required for termination in human cells. EMBO Rep, e56150, doi:10.15252/embr.202256150 (2023).
8 Maqbool, M. A. et al. Alternative Enhancer Usage and Targeted Polycomb Marking Hallmark Promoter Choice during T Cell Differentiation. Cell Rep 32, 108048, doi:10.1016/j.celrep.2020.108048 (2020).



