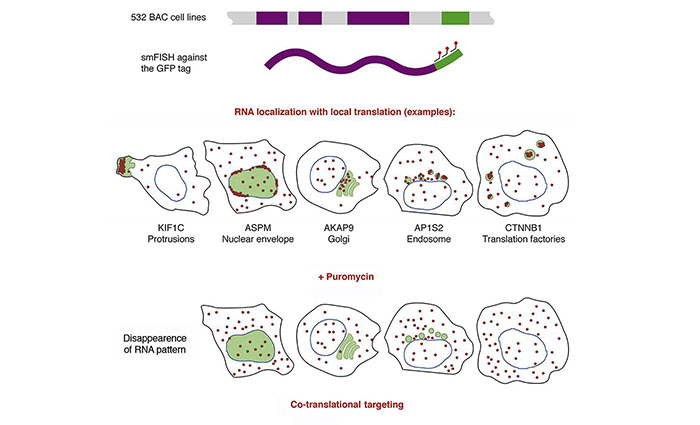
La traduction locale permet un contrôle spatial de l’expression des gènes. Dans cette étude, nous avons effectué un double criblage de localisation protéine-ARNm, en couplant une détection par FISH en molécule unique de l’ARN sur 523 lignées cellulaires humaines exprimant des gènes marqués par la GFP. 32 ARNm présentaient des localisations cytoplasmiques spécifiques avec une traduction locale à des endroits inattendus, y compris les protrusions cytoplasmiques, les bords des cellules, les endosomes, le Golgi, l’enveloppe nucléaire et les centrosomes, ces derniers étant dépendant du cycle cellulaire. La classification automatisée des modèles de localisation de l’ARNm a révélé un degré élevé d’hétérogénéité intercellulaire. De manière surprenante, la localisation de l’ARNm nécessitait fréquemment une traduction en cours, indiquant un ciblage d’ARN co-traductionnel répandu. Fait intéressant, alors que l’accumulation dans les P-bodies était fréquente (15 ARNm), quatre ARNm se sont accumulés dans des foyers qui étaient des structures distinctes. Ces foyers manquaient de protéine mature, mais l’imagerie des polypeptides naissants a montré qu’il s’agissait d’usines de traduction spécialisées. Pour la β-caténine, la formation des foyers était régulée par Wnt, reposait sur l’agrégation de polysomes dépendant d’APC et conduisait à une dégradation des protéines naissantes. Ainsi, les usines de traduction régulent de manière unique le métabolisme des protéines naissantes et créent une compartimentation granulaire fine de la traduction.
Racha Chouaib 1, Adham Safieddine 1, Xavier Pichon 1, Arthur Imbert 1, Oh Sung Kwon, Aubin Samacoits, Abdel-Meneem Traboulsi, Marie-Cécile Robert, Nikolay Tsanov, Emeline Coleno, Ina Poser, Christophe Zimmer, Anthony Hyman, Hervé Le Hir, Kazem Zibara, Marion Peter, Florian Mueller*, Thomas Walter*, Edouard Bertrand*
DOI:https://doi.org/10.1016/j.devcel.2020.07.010
Article mis en évidence dans Dev Cell par Ashley Chin & Eric Lécuyer.
Translating Messages in Different Neighborhoods
DOI:https://doi-org.insb.bib.cnrs.fr/10.1016/j.devcel.2020.09.006
Egalement sur la même thématique au sein de l’équipe Edouard Bertrand IGMM et disponible sur BioRxiv :
Adham Safieddine, Emeline Coleno, Abdel-Meneem Traboulsi, Oh Sung Kwon, Frederic Lionneton, Virginie Georget, Marie-Cécile Robert, Thierry Gostan, Charles Lecellier, Soha Salloum, Racha Chouaib, Xavier Pichon, Hervé Le Hir, Kazem Zibara, Marion Peter, Edouard Bertrand