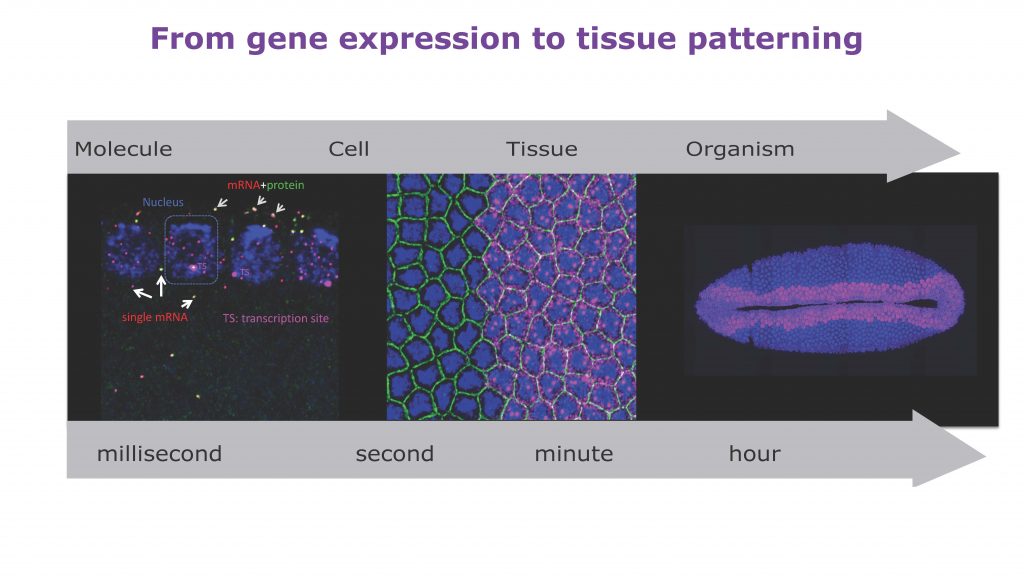
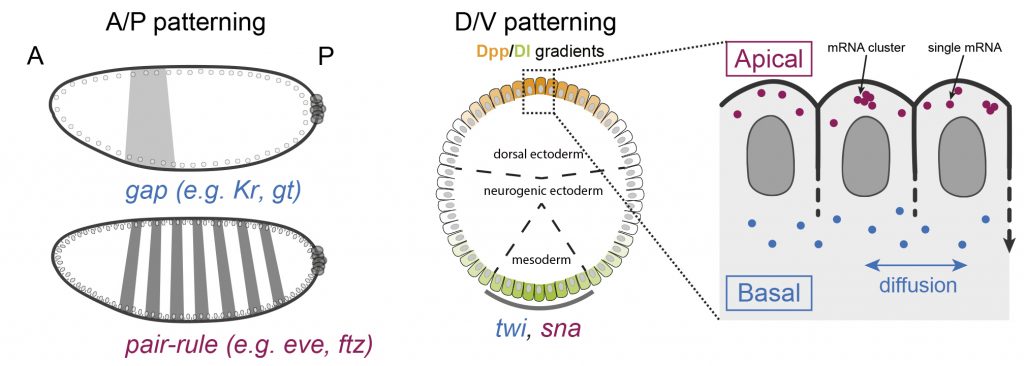
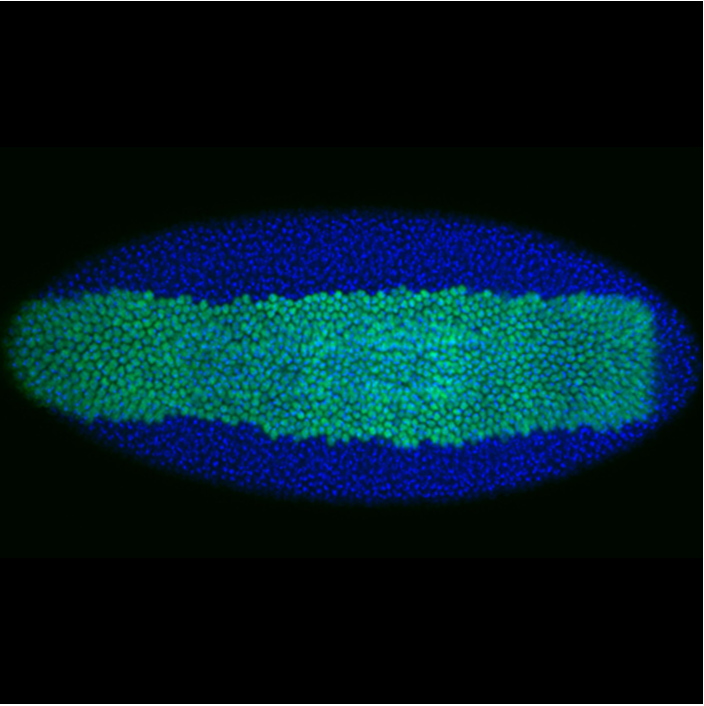
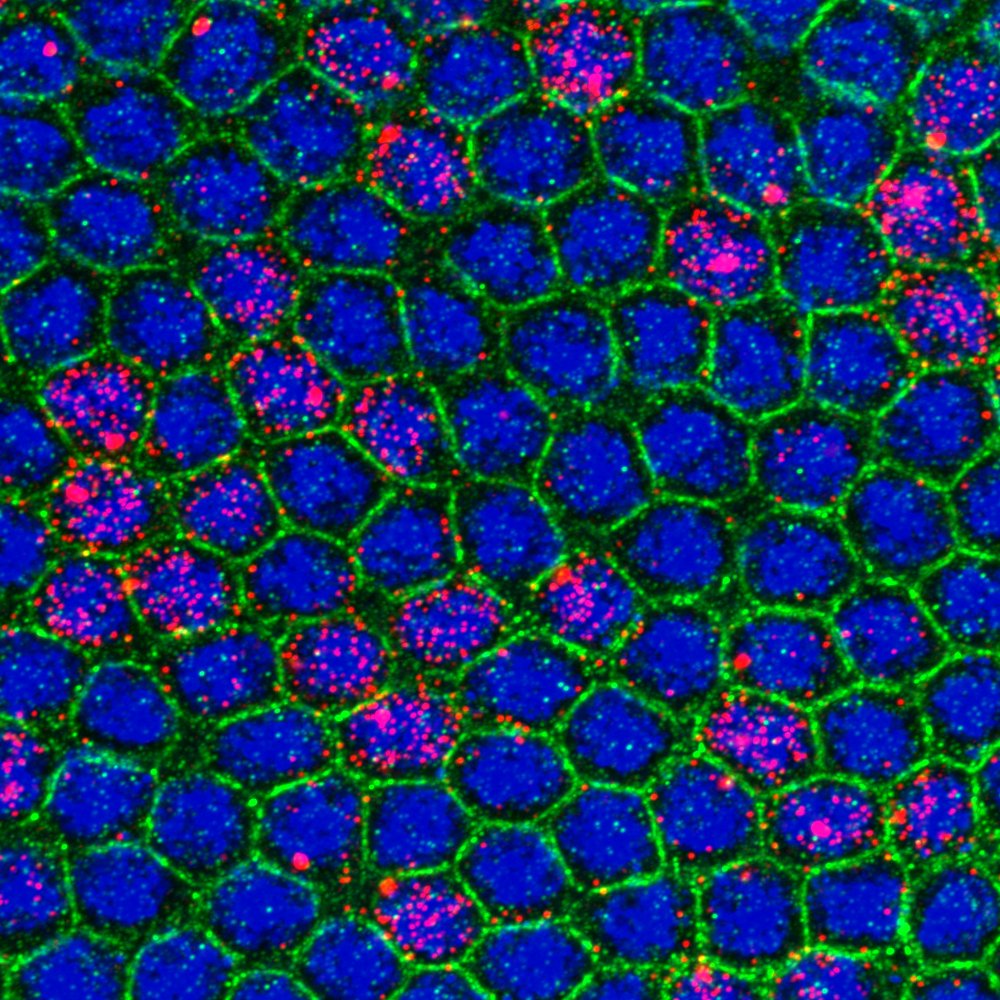
Le développement harmonieux d’un organisme pluricellulaire nécessite un contrôle de l’expression de ses gènes afin que les cellules puissent adopter un destin précis dans l’espace et dans le temps. Nos recherches visent à décrypter les mécanismes permettant d’atteindre une telle précision. Nous nous intéressons aux aspects temporels des deux étapes clés de la régulation de l’expression du génome: la transcription, étape de photocopie de l’information codée dans notre génome en une molécule intermédiaire appelée acide ribonucleique messager (ARNm) et la traduction, processus de formation de protéines à partir de ces ARNm. Afin d’étudier ces processus, nous utilisons l’embryon de drosophile, organisme modèle de choix pour les manipulations génétiques et où il est aisé de suivre en temps réel le réveil du génome.
En combinant de l’imagerie quantitative sur embryons vivants avec de la modélisation, notre équipe a pu décoder le rôle des séquences régulatrices du génome dans la coordination temporelle de l’activation des gènes et son héritabilité entre les cellules mères et leurs descendantes. Par ailleurs, nous avons réussi à visualiser où et quand des molécules uniques d’ARNm sont traduites dans un embryon. Cette avancée technologique a révélé une surprenante variabilité dans l’efficacité de traduction des ARNm en fonction de leur localisation dans la cellule. Dans le futur, nous projetons de comprendre plus largement la fonction de la localisation des ARNm et leurs conséquences sur la traduction. Par ailleurs, nous souhaitons étudier comment la vitesse d’expression de notre code génétique peut directement influencer la production de protéines. Dans les cellules eucaryotes, transcription et traduction s ‘effectuent dans deux compartiments cellulaires distincts et sont de ce fait généralement envisagées comme des processus indépendants.
Nous questionnerons cette indépendance grâce à de la microscopie à haute résolution dans un organisme vivant.
PROJECT 1
How do translation dynamics of localized patterning transcripts modulate cell fate decisions?