Nos recherches visent à comprendre comment l’organisation supérieure de la chromatine est impliquées dans le contrôle de l’expression des gènes chez les mammifères, dans des situations normales ou pathologiques.
En contexte physiologique, grâce au développement de techniques expérimentales et d'outils de modélisation originaux, notre équipe s’intéresse aux propriétés de l'organisation de la chromatine à deux niveaux importants : les domaines d'association topologique (TADs) (Axe 1) et les compartiments nucléaires en lien avec les corps nucléaires (Axe 2).
En contexte pathologique, nous nous intéressons à la régulation du gène SNCA codant l’α-synucléine, protéine responsable de la maladie de Parkinson (Axe 3).
1.Coordination de l’expression des gènes chez les mammifères à l’intérieur des TADs
Notre équipe a contribué au développement des technologies de Capture de Conformation de Chromosome (3C) en améliorant la sensibilité des tests de 3C (méthode 3C-qPCR) (Hagège et al., Nature Protocols 2, 1722, 2007; Rebouissou et al., Meth. Mol. Biol. 2532, 187-197, 2022). Grâce à l’utilisation de de cette méthode, nous avons montré que les loci riches en gènes possèdent des fréquences de contact modulées périodiquement en fonction de la distance séparant les sites le long du génome (Court et al., Genome Biol. 12, R42, 2011). Après avoir initié une collaboration interdisciplinaire avec Annick Lesne, physicienne, nous avons montré que cette modulation peut être décrite par des modèles dérivés de la physique des polymères, comme si la chromatine était repliée statistiquement en forme d’hélice (modèle d’hélice statistique) (Ea et al., Genes 6, 734-750, 2015). Nous avons ensuite montré que, en raison de leur taille différente, des principes de physique des polymères différents régissent la dynamique de la chromatine dans les domaines topologiques de la souris et de la mouche Drosophile (Ea et al., BMC Genomics 16, 607, 2015; collab. avec G. Cavalli, IGH, Montpellier).
Plus récemment, nous avons montré que certaines séquences rétrovirales endogènes (ERV) agissent dans le génome de la souris comme des activateurs transcriptionnels (« enhancers ») putatifs contrôlant l'expression de gènes endogènes par le biais d'interactions chromatiniennes à longue distance contrôlées par HP1 (Heterochromatin Protein 1) (Calvet et al., Cells 11, 2392, 2022 ; collab. avec F. Cammas, IRCM, Montpellier).
Enfin, dans une étude in-silico, grâce à l’utilisation de données d’association pangénomique (GWAS), nous avons montré que, pour une fraction des maladies humaines, les variants nucléotidiques (SNPs) à risque sont préférentiellement localisés dans les bordures de domaines topologiques (TADs) (Jablonski et al., Human Genomics, 16, 2, 2022; collab. avec M.T. Hütt, Brême, Allemagne). Les cancers sont relativement plus fréquents parmi ces maladies et, grâce au soutien de La Fondation ARC, nous caractérisons maintenant l’organisation 3D de la chromatine d’un locus paradigmatique associé à un risque élevé de cancer du poumon.
2.Corps nucléaires et organisation supérieure de la chromatine chez les mammifères
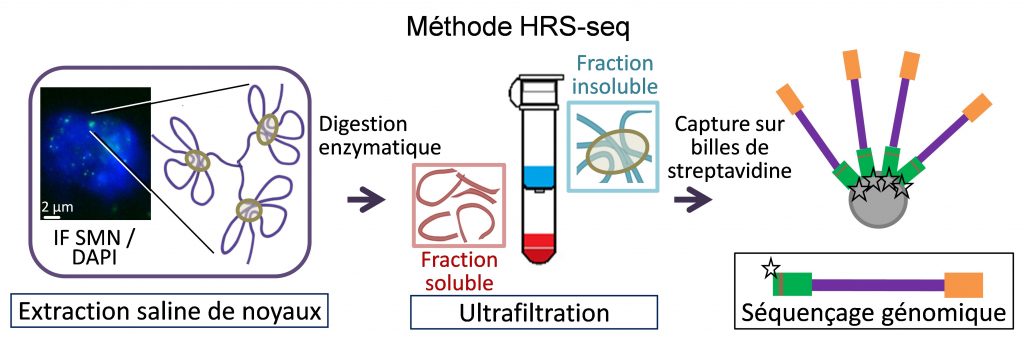
Les corps nucléaires sont des organelles nucléaires non-membranaires qui se comportent essentiellement comme des gouttelettes liquides. Beaucoup d’entre eux s’assemblent in vivo par des processus de séparation de phase et jouent un rôle essentiel dans la coordination de l’expression des gènes en confinant ensemble des régions chromatiniennes spécifiques dispersées dans le génome (Lesne et al., Genes 10, 1049, 2019). Nous avons développé une nouvelle méthode, appelée HRS-seq (High-salt Recovered Sequences-sequencing), permettant l’analyse génomique par séquençage à haut-débit des régions associées aux larges complexes ribonucléoprotéiques (RNPs) du noyau incluant plusieurs types de corps nucléaires. Dans les cellules souches embryonnaires (ES) de souris, nous avons montré que le compartiment chromosomique actif est associé à de tels complexes RNPs (Baudement et al., Genome Res. 28, 1733-1746, 2018; collab. avec L. Journot, IGF, Montpellier et J. Mozziconacci, MNHN, Paris ).
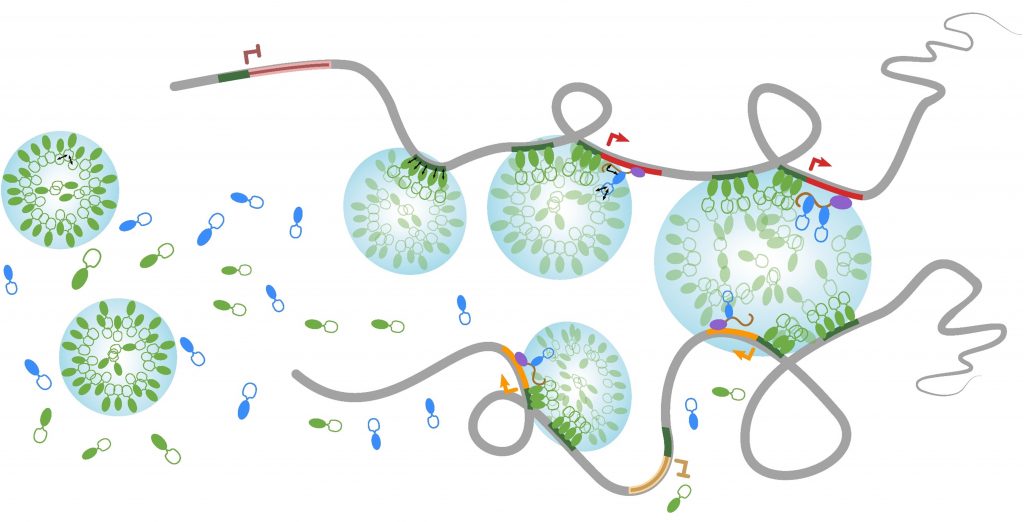
Nous explorons maintenant le rôle que ces corps nucléaires jouent dans l'organisation supérieure de la chromatine et dans le contrôle de l'expression des gènes, pendant la différenciation cellulaire ou dans certaines situations pathologiques. Afin d'identifier plus précisément les gènes associés à une classe spécifique de corps nucléaires, nous travaillons à adapter la méthode HRS-seq en agissant sur les paramètres physiques de la séparation de phase (projet ARGECOR 80Prime, soutenu par la mission interdisciplinaire du CNRS - MITI).
3.Régulation transcriptionnelle de l’expression de l’α-synucléine dans la maladie de Parkinson
La maladie de Parkinson, dont les symptômes moteurs sont principalement dus à la disparition sélective des neurones dopaminergiques de la Substance Noire pars compacta (SNpc), est la deuxième maladie neurodégénérative la plus courante chez l’Homme. La majorité des cas résultent d’une combinaison de facteurs génétiques et environnementaux. Le gène SNCA qui code pour la protéine α-synucléine, une protéine neuronale très présente dans les terminaisons présynaptiques et jouant un rôle dans la libération de neurotransmetteurs, est le premier gène à avoir été associé à la maladie de Parkinson. Une augmentation même limitée de l'expression d'α-synucléine de type sauvage peut provoquer des formes familiales ou sporadiques de la maladie de Parkinson. Plusieurs variants nucléotidiques (SNPs) du locus SNCA, identifiés dans les études d'association pangénomique (GWAS) comme étant fortement associés à un risque accru de la maladie de Parkinson, corrèlent avec des niveaux plus élevés d'α-synucléine chez les patients. La réduction de l'expression d'α-synucléine représente donc une stratégie thérapeutique intéressante. Pour cela, une connaissance approfondie des mécanismes moléculaires impliqués dans la régulation transcriptionnelle de SNCA en condition normale, mais surtout en conditions pathologiques, est nécessaire.
Grâce au soutien de la Fondation pour la Recherche Médicale (FRM), nous cherchons à identifier les mécanismes d’activation du gène SNCA dans des conditions normales et pathologiques. Pour cela, nous utilisons des modèles cellulaires représentant les formes sporadiques (cellules LUHMES cultivées en 3D, différenciées en neurones dopaminergiques et traitées au MPP+) ou familiales (organoïdes dérivés de cellules pluripotentes induites portant la mutation p.A53T de l’α-synucléine) de la maladie de Parkinson. A l’aide de ces modèles, nous tentons de caractériser les éléments génomiques clés de régulation et les facteurs de transcription responsables de l'induction pathologique de SNCA. Pour cela, en collaboration avec Solange Desagher (IRIM, Montpellier), Franck Court (GReD, Clermont-Ferrand) et de Philippe Ravassard (ICM, Paris), nous combinons des approches basées sur l’architecture 3D de la chromatine (HRS, 4Cseq) avec les données GWAS disponibles, la protéomique et l'édition du génome par CRISPR-Cas9.


