Notre équipe étudie l’épigénétique chez les mammifères et plus particulièrement les mécanismes épigénétiques qui contrôlent les gènes soumis à l’empreinte. Ces gènes peu communs sont organisés en domaines chromosomiques évolutivement conservés, et jouent divers rôles. Ils sont essentiels dans le développement et dans certaines maladies. Les gènes soumis à l’empreinte sont exprimés d’une manière mono-allélique, dépendant uniquement de la provenance parentale du gène, soit du père ou de la mère. Cette expression allélique est contrôlée par empreinte génétique due à une méthylation différente de l’ADN (« methylation imprints ») sur des séquences régulatrices clés, mais dépend aussi d’autres caractéristiques de la chromatine.
Une des thématiques de recherche de notre laboratoire est la compréhension de l’établissement de l’empreinte épigénétique dans la lignée germinale et sa maintenance dans les cellules somatiques. En lien avec ce thème, nous étudions comment la méthylation mono-allélique entraîne l’expression de gène soumis à l’empreinte dans l’embryon, souvent dans des tissus spécifiques. Les résultats préliminaires de nos recherches ont identifié l’implication essentielle des longs ARNs non codants (lncRNAs) et un changement de méthylation des histones dans des domaines soumis à l'empreinte.
Ces dernières années notre laboratoire s'est intéressé à un second grand thème : l’exploration de la structuration des domaines chromosomiques dans le noyau en utilisant de nouvelles technologies. Nous nous sommes penchés sur les questions suivantes : est-ce que la chromatine des domaines soumis à l’empreinte présente une structure différente sur les chromosomes maternels par rapport aux paternels ? Est-ce que cette différence de structure contribue à l’expression mono allélique des gènes soumis à l’empreinte ? Pour répondre à ces questions nous avons utilisé des approches de microscopie avancées pour observer la compaction et la structuration de la chromatine à l’échelle nanométrique dans des cellules vivantes. Cette nouvelle étude basée sur le FRET a permis de déchiffrer les rôles structuraux de régulateurs de la chromatine en complément d’approche ciblant les gènes dans la souris et le nématode. Les études combinées de notre laboratoire ont pour but d’améliorer notre compréhension de la régulation des gènes soumis à l’empreinte. Et d’étudier leur contribution dans des maladies humaines spécifiques.
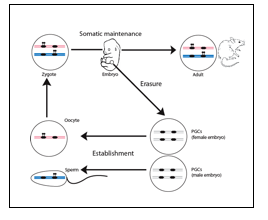
Les mécanismes épigénétiques jouent divers rôles dans le développement et définissent de façon stable la répression ou l’expression des gènes dans des lignées cellulaires spécifiques. Chez les mammifères, il y a plusieurs exemples d’inactivations somatiques héritables sur un des deux allèles d’un gène. Cela inclut les gènes présents sur le chromosome X chez les femelles (inactivation du X) et les gènes soumis à l’empreinte, un ensemble indispensable de 150 gènes codants pour des protéines (et des centaines codant pour des RNA non codants de régulation). L’expression des gènes soumis à l’empreinte dépend entièrement du fait que le gène soit hérité de la mère ou du père, et joue un rôle dans la prolifération cellulaire, l’homéostasie et le comportement. L’expression mono-allélique des gènes soumis à l’empreinte est sous l'influence des régions de contrôle de l’empreinte (ICRs pour ‘Imprinting Control Regions’). Elles sont essentiellement composées de séquences riches en GC de plusieurs kilobases, sur lesquelles les marques épigénétiques sont déposées en fonction de leur passage dans la lignée germinale autant femelle que mâle. La nature précise de ces empreintes génétiques parentale n’est pas encore complètement comprise, mais elles sont caractérisées par une méthylation de l’ADN. L’établissement de l’empreinte par une méthylation mâle ou femelle spécifique nécessite l'action de la méthyltransférase de novo Dnmt3a et de son cofacteur Dnmt3-like (Dnmt3l). Après fécondation, les empreintes de méthylation maternelles et paternelles sont maintenues à travers le développement dans toutes les cellules somatiques (figure 1). La dérégulation de ces processus essentiels est impliquée dans les retards de croissance fœtale du syndrome de Silver-Russell (SRS). Cette dérégulation est aussi impliquée dans le syndrome de Beckwith-Wiedemann (BWS), le diabète néonatal transitoire (TNDM) mais aussi dans divers troubles neuro-comportementaux. Tout comme les gènes suppresseurs de tumeurs, la méthylation de l’ADN est souvent perturbée dans le cancer sur les locus soumis à l’empreinte et contribue à la cancérogénèse.
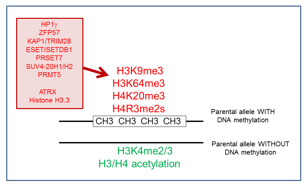
Figure 1: Contrôle développemental de la méthylation de l’ADN régulant l’expression allélique des gènes soumis à l’empreinte. Les hypothétiques empreintes génomiques par méthylation sont figurées (pastilles noires). Le génome maternel est présenté en rose, le paternel en bleu. Dans les cellules germinales primordiales (PGCs), l’empreinte est effacée pour permettre un « nouveau cycle d’empreinte » pour la nouvelle génération.
Adapté d'Hirasawa & Feil, BioEssays 2010.
1.Maintenance de l’empreinte parentale durant le développement.
Dans cette thématique, nous nous sommes concentrés sur l'étude des empreintes par méthylation de l’ADN mono-allélique sur les ICRs et leur maintien durant le développement, plus particulièrement durant les étapes critiques avant l’implantation. Notre équipe a étudié la présence et l’importance de la méthylation des lysines et arginines sur les histones au niveau des ICRs, qui présente une combinaison de marques différente entre l’allèle méthylé et non méthylé (figure 2). Plus récemment, nous nous sommes intéressés au rôle de facteurs de transcription spécifiques. Les études utilisent la souris comme modèle pour étudier différents domaines soumis à l’empreinte et leur régulation épigénétique à l’échelle du génome. En lien avec l’exploration de la chromatine au niveau des ICRs et comment la différence de méthylation est régulée tout au long du développement, la question de leur origine évolutive et leur importance est également intéressante. Ce thème est notamment approprié pour comprendre les pathologies humaines dans lesquelles la perte ou le gain de méthylation de l’ADN sont souvent impliqués. C’est pourquoi nous avons établi des collaborations avec des groupes cliniques pour explorer la conservation de ces mécanismes sous-jacents chez l’humain et explorer leurs importances dans les maladies liées à l’empreinte.
Recent publications on this research theme
Henckel A., Chebli K., Kota S.K., Arnaud P., and Feil R. (2012). Transcription and histone methylation changes correlate with imprint acquisition in male germ cells. EMBO Journal, 31, 606-615.
Girardot M., Hirasawa, R., Kacem, S., Fritsch, L., Pontis, J., Kota, S.K., Filipponi, D., Fabbrizio, E., Sardet, C., Lohmann, F., Kadam, S., Ait-Si-Ali, S. and Feil, R. (2014). PRMT5-mediated histone H4 arginine-3 symmetrical dimethylation marks chromatin at G+C rich regions of the mouse genome. Nucleic Acids Research, 42, 235-248.
Auclair, G., Borgel, J., Sanz, L.A., Vallet, J., Guibert, S., Dumas, M., Cavelier, P., Girardot, M., Forné, T., Feil, R.* and Weber, M.* (2016). EHMT2 directs DNA methylation for efficient gene silencing in mouse embryos. Genome Res, 26, 192-202.
Prats-Puig, A., Carreras-Badoso, G., Bassols, J., Cavelier, P., Magret, A., Sabench, C., de Zegher, F., Ibanez, L., Feil, R.*, Lopez-Bermejo, A.* (2017). The placental imprinted DLK1-DIO3 domain: a new link to pre- and postnatal growth in human beings. Am. J. Obstet. & Gynecology, Epub before print.
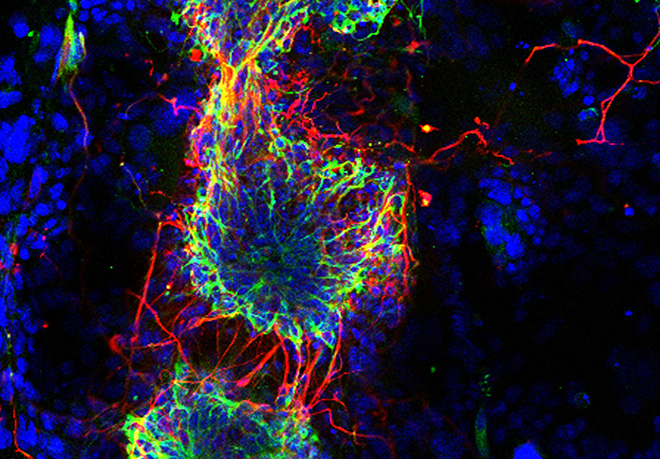
Figure 2: Organisation différente de la chromatine aux régions de contrôle de l’empreinte ICRs dans les cellules somatiques. Sur l’allèle parental marqué de l’empreinte par la méthylation de l’ADN, il y a une méthylation répressive des histones (en rouge) régulée par différentes protéines (affichées à gauche). L’allèle opposé non méthylé est marqué par des modifications de ‘chromatine active’ (en vert). (Sanli et al, Int. Biochem. Cell Biol. 2015)
(Sanli et al., Int J. Biochem. Cell Biol. 2015).
2.Comment les ICRs engendrent-ils l’expression des gènes soumis à l’empreinte en cis ?
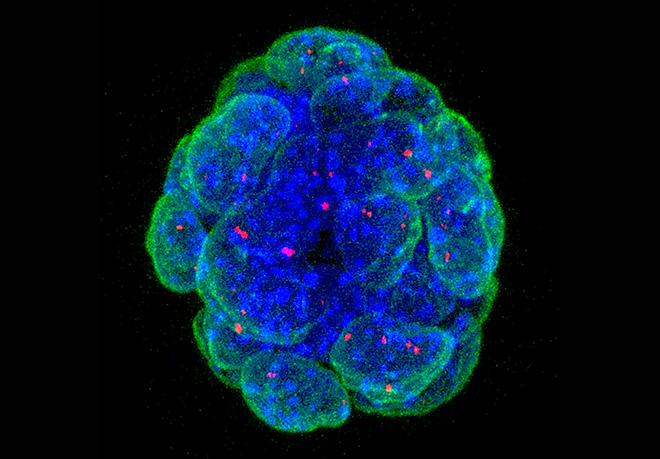
Cela a été une thématique longtemps étudié dans le laboratoire et notre axe de recherche adresse la question de l'apport de la méthylation de l’ADN parentale dépendante sur les ilots CpG sur l'expression mono-allélique de gène sur des locus soumis à l’empreinte. Ce processus encore peu compris se produit durant l’embryogenèse, souvent de manière lignage spécifique. Dans nos travaux précédents, nous avons montré l’importance de la méthylation des lysines des histones dans le contrôle développemental de l’expression de gène soumis à l’empreinte. Plus particulièrement, nous avons reporté divers rôles de l’histone méthyltransférase G9A (EHMT2) et du Polycomb répressive Complex 2 (PRC2). Sur plusieurs domaines soumis à l’empreinte, l’ICR produit des long ARNs non codants (lncRNA) qui sont retenus dans le noyau et altèrent l‘expression des gènes en cis. Pour adresser plus en détail ces mécanismes d’action, nous avons étudié le domaine soumis à l’empreinte Dlk1-Dio3 sur le chromosome murin 12. Nous avons trouvé que l’ICR intergénique de ce domaine agit comme un « enhancer », contrôlant en cis l’expression allélique de plusieurs ncRNA. Un de ces ncRNA maternelle est suspecté de réprimer en cis des gènes codants pour des protéines de ce large domaine. De manière intéressante, ce lncRNA appelé Meg3 (‘Maternally expressed gene-3’) est séquestré sur son site de transcription. Nous avons développé différentes manières de visualiser Meg3 dans le noyau (figure 3) en se basant sur l’approche de marquage MS2 et la détection RNA par FISH de la molécule unique.
Recent publications on this research theme
Sanli, I.*, Lalevée, S.*, Camissa, M., Perrin, A., Rage, F., Riccio, A., Llères, D., Bertrand, E., Feil, R. (2018). Meg3 long non-coding RNA expression controls imprinting by preventing transcriptional upregulation in cis. Cell reports, 23, 337-348.
Kota, S.K.*, Lleres, D.*, Bouschet, T., Hirasawa, R., Marchand, A., Begon-Pescia, C., Sanli, I, Arnaud, P., Journot, L., Girardot, M. and Feil, R. (2014). ICR non-coding RNA expression controls imprinting and DNA replication at the Dlk1-Dio3 domain. Dev Cell, 31, 19-33.
Puget, N., Hirasawa, R., Nguyen Hu, N-S., Laviolette-Malirat, N., Feil, R.* & Khamlichi, A.A.* (2015). Insertion of an imprinted insulator into the IgH locus reveals developmentally regulated, transcription-dependent suppression of V(D)J recombination. Mol Cell Biol. 35, 529-543.
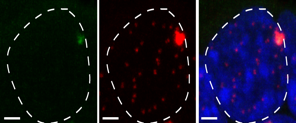
Figure 3: FISH detection of the Meg3 long non-coding RNA of the Dlk1-Dio3 imprinted domain in ES cells, using a classical cDNA probe (green) or fluorescent oligonucleotides that detect the inserted MS2 repeats (red). The image to the right shows the overlay. Scale bar, 5 mm
(Sanli/Lalevée et al., Cell reports 2018)
3.Organisation nucléaire des domaines de gènes soumis à l’empreinte.
La façon dont les domaines chromosomiques soumis à l’empreinte sont organisés dans l’espace à l’intérieur du noyau reste encore peu connue, ainsi que les différences d’organisation structurale entre le génome maternel et paternel. Une question en lien avec ces inconnues est de savoir si l’organisation d’ordre supérieur des domaines soumis à l’empreinte est régulée lors du développement, et peut donc contribuer à une expression génique tissue spécifique. Afin d’étudier la topologie nucléaire des domaines, nous avons choisi le locus Dlk1-Dio3 sur le chromosome 12 de la souris comme locus modèle ainsi que d’autres locus soumis à l’empreinte. Dans nos études sur des cellules souches embryonnaires (ES) et différenciées, nous avons utilisé des approches d’hybridation in situ (FISH) et, en collaboration avec d’autres laboratoires, appliqué des techniques de capture de conformation de chromosome (4C). Ces approches combinées nous ont fourni d’importantes connaissances sur les différences de structures entre les chromosomes maternels et paternels, et sur leur différence de contribution lors de l’expression allélique des gènes soumis à l’empreinte.
Recent publications on this research theme
Kota, S.K.*, Lleres, D.*, Bouschet, T., Hirasawa, R., Marchand, A., Begon-Pescia, C., Sanli, I, Arnaud, P., Journot, L., Girardot, M. and Feil, R. (2014). ICR non-coding RNA expression controls imprinting and DNA replication at the Dlk1-Dio3 domain. Dev Cell, 31, 19-33.
Sanli, I. and Feil, R. (2016). PRC-mediated interaction networks of repressed genes: emerging insights and possible roles. Epigenomics, 8, 733-735.
4.Étude de la structure de la chromatine à l’échelle nanométrique dans des cellules et animaux vivants.
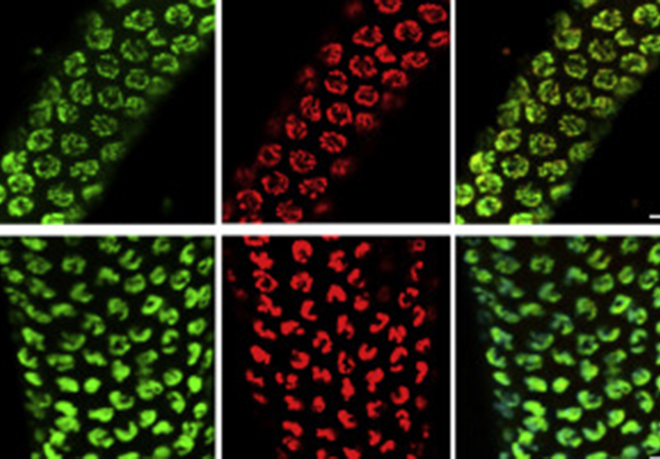
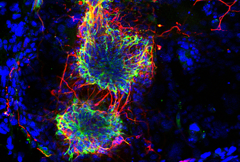
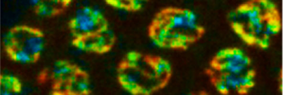
Nous avons récemment adapté une approche de microscope basée sur le FRET pour mesurer la compaction de la chromatine dans des cellules vivantes. Plus particulièrement, cette nouvelle approche basée sur la mesure non invasive du temps de vie de fluorescence (FLIM) nous permet de suivre la structure et la compaction de la chromatine à l’échelle nanométrique. Nous avons avec succès utilisé cette approche de microscopie quantitative sur des nématodes vivants. Nous avons défini des états de compaction de la chromatine tout le long de chromosomes méiotique, et quel était le rôle des complexes de condensines lors des phases spécifiques de la méiose. Cette approche a également été réalisée dans des cellules cancéreuses, ce qui nous a permis d’étudier les rôles structuraux de protéines spécifiques associées à la chromatine. Pour l’avenir, nous avons pour but d’utiliser cette approche dans des cellules primaires de souris et, d’explorer le rôle de modificateur de la chromatine à l’échelle nanométrique. Une autre perspective excitante est l'exploration des effets génétiques et environnementaux sur la compaction de la chromatine.
Recent publications on this research theme
Lleres, D.*, Bailly, A.P.*, Perrin, A., Norman, D., Xirodimas, D.P., Feil, R. (2017). Quantitative FLIM-FRET microscopy to monitor nanoscale chromatin compaction in vivo reveals structural roles of condensin complexes. Cell reports, 18, 1791-1803.
Sobecki M, Camasses A, Mrouj K, Parisis N, Nicolas E, Lleres D, Gerbe F, Prieto S, Krasinska L, David A, Egure M, Birling M-C, Hem S, Dejardin J, Malumbres M, Jay P, Dulic V, Lafontaine DLJ, Feil R, Fisher D (2016). Ki-67 organises heterochromatin and controls gene expression during cell proliferation. ELife, e13722.